RNA测序遇上Dup,有你想不到的误区 | BGISEQ平台低Dup rate优势全解
【知识卡片】什么是Dup?Dup,即重复序列Duplicate reads,指通过测序得到两对或两对以上的Pair-End Reads,同时比对到参考基因组上相同的起始和结束位置的序列。这些重复序列在总测序序列中的占比简称为Dup rate。
Dup会影响DNA测序变异检测结果准确性,需要在生信分析中去除,只是会产生测序成本浪费。RNA测序遇上Dup,问题就更复杂了。一类Dup是建库或测序过程引入的“坏”Dup,另一类Dup是样本高基因表达形成相同模板的“好”Dup。
如果不分“好”“坏“全去,就会损失这部分“好”的数据;如果不去,Dup会影响RNA测序基因表达定量的准确性。如何识别和去除RNA测序“坏”Dup,一直是一个热点,众说纷纭,方法各异。
01、Dup如何影响RNA测序?
相关文献报道:
(1)高Dup rate会影响差异表达基因的检测。
Sergi Sayols等人[1]从数据库获取RNA-Seq数据,模拟PCR Dup rate 50%和90%。Dup rate 50%时,能还原出原数据1199个差异表达基因,产生124个假阳性和720个假阴性;Dup rate 90%时,问题加重,只还原出517个,产生115个假阳性和1402个假阴性(图1 D-F)。
(2)对于低表达基因,Dup rate偏移程度更大。
Sergi Sayols等人[1]还分析了不同Dup rate程度下,低表达基因的偏移。本来低表达基因表现低Dup rate,随着基因表达水平升高,Dup rate升高。但在高Dup rate的情况下,低表达基因已经出现了高Dup rate(图1 A-C)。
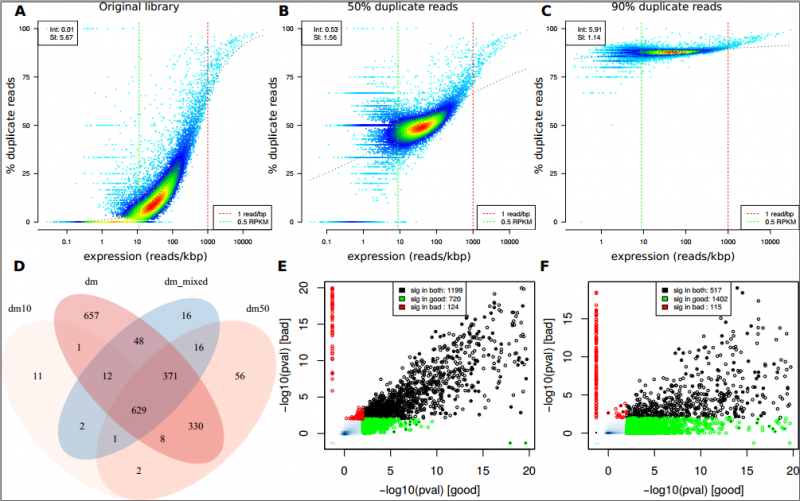
图1 不同Dup rate程度,对同一文库中检测差异表达基因的影响
(3)对于较短的基因,打断时更易产生相同的reads,被当作Dup。
Yu Fu等人[2]指出相同的表达水平下,短的基因比长的基因,reads更可能被当成Dup,因为在建库时有一步骤是将RNA随机片段化,对短的基因来说打断成不同片段的“空间”更小。
02、PCR Dup是干扰RNA测序的“罪魁祸首”吗?
RNA测序Dup是怎么产生的?来源有以下4个:
(1)样本本身的Dup;
(2)文库构建中扩增引入的Dup(即PCR Dup);
(3)测序前信号放大(荧光信号采集单元生成过程)引入的Dup;
(4)芯片测序过程中引入的光学Dup。
大多数人认为Dup主要来自于第2种,即建库环节中引入的PCR Dup,因而目前的文章中以降低建库环节引入的PCR Dup为主,或在生信环节通过开发软件,识别出PCR Dup;或在建库环节通过加分子标签,依据带有相同标签判断为PCR Dup。
PCR Dup真的是“罪魁祸首”吗?此前的文章(《NGS攻略》之Dup传)详细地剖析了前人文章和实测数据,分辨每种Dup的“影响力”。PCR Dup在扩增循环数目不高的情况下,并不会带来“令人窒息”的Dup rate,而只是在个位数水平波动。实际上第3种和第4种,即测序引入的Dup居多。
03、测序引入的Dup rate有多大?
从上百个商业样本DNA测序看,BGISEQ和H平台Dup rate的差距明显,BGISEQ平台在3%以下,H平台>20%(Duplicates | NGS帝国的Agent Smith)。RNA测序表现如何呢?
1. 人标准品数据
(1)真核转录组测序
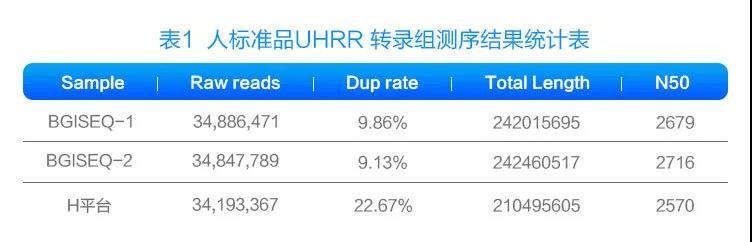
人标准品UHRR构建真核转录组文库,分别在BGISEQ和H平台上机,测序策略PE150。BGISEQ Dup rate,仅有9%左右, H平台高达22.67%。(表1)。
(2)lncRNA测序
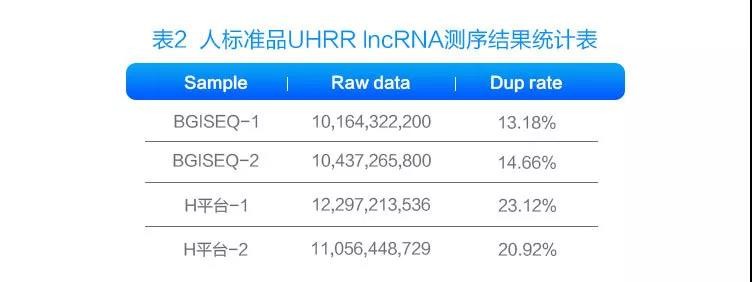
人标准品UHRR构建lncRNA文库,分别在BIGSEQ和H平台上机,测序策略PE100。BGISEQ Dup rate,仅有13%左右,H平台超过20%(表2)。
2. 商业项目统计
统计近期BGISEQ PE150测序的真核转录组商业项目,涉及多种植物、动物和真菌共152个样本,平均Dup rate仅为10.36%,而H平台29个样本,平均28.74%。
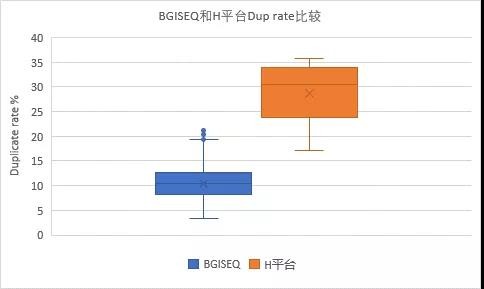
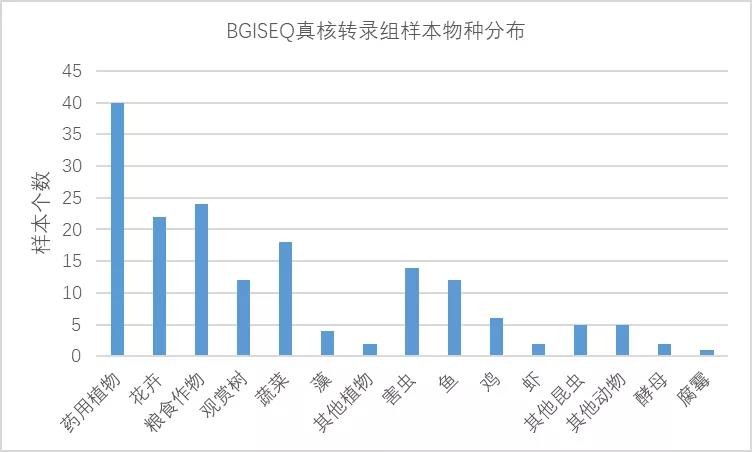
图2 近期BGISEQ真核转录组商业项目情况
从RNA测序数据中不难发现,单看BGISEQ平台结果,4种Dup来源加一起也不过百分之十,而两个平台间就差了百分之十几。可见测序引入的Dup比其他Dup多得多,BGISEQ平台Dup rate很低。
04、BGISEQ Dup rate为什么这么低?
这得从原理上说起,BGISEQ平台基于独特的核心技术,减少了测序环节引入的Dup,使得Dup rate明显低于H平台。
原因1:BGISEQ平台采用DNB技术,PCR双链文库在后续环化实验中只环化双链中的1条,滚环扩增获得DNA Nanoball;另外一条则被核酸外切酶消化,不用于形成DNA Nanoball,因此是单链的模板。
而H平台,是双链PCR产物变性与芯片的接头互补连接,双链中的两条链是一样的信息,都可以在后面桥式扩增中作为模板,这样引入的Dup比BGISEQ平台高。此外,BGISEQ平台是线性扩增,始终只复制原始模版,其扩增错误不会像H平台桥式PCR一样累积成指数型放大。
原因2:BGISEQ平台采用Patterned Array技术,制备好的DNB会加载到微阵列芯片(Patterned Array)上,这一过程称为DNB加载。Patterned Array技术通过先进的半导体精密加工工艺,在硅片表面形成阵列和对准标记,因而芯片上活化位点是规则阵列的,而且DNB与芯片上活化位点的大小相近,每个位点只固定一个DNB,保证信号点之间不产生相互干扰。再结合高分辨率成像系统和自主开发算法等提高了图像处理精准度,大大提高了碱基识别准确度。
而H平台有两种芯片,一种是较早系列,采用非阵列式芯片,生成的DNA cluster形状不规则,图像识别有可能把一个cluster识别成两个,会造成Dup的问题;另一种是近年来的超大通量测序平台,为了更多有效数据,采用阵列式芯片,在进行第一条互补链合成后,DNA模板分子会脱落,并有一定的概率到达另一个纳米孔生成另一个相同的cluster,造成Dup的问题。
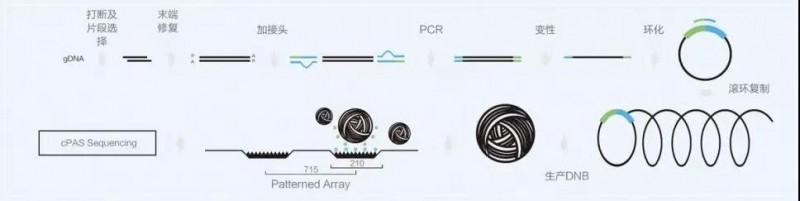
图3 BGISEQ平台原理图
BGISEQ平台独具低Dup rate先天优势,就无需在生信环节人为地设计软件去除,也免去建库环节费尽心力地人为干预样品建库,省时省力省钱。
BGISEQ RNA测序产品亮点颇多,除了低Dup rate,还有:
11天极速交付:BGISEQ真核转录组和RNA-Seq,提取检测为起始,有参无参都极速。提取2天;打包服务包含检测,RNA-Seq 只需要11天,真核转录组只需要14天。
“0”index hopping:独特的文库构建技术和单链环状文库滚环扩增技术使得index hopping在0.0001%~0.0004%,远低于H平台[3]。
Dr. Tom 2.0多组学:任意测mRNA/lncRNA/Small RNA一种,获取多组学的关联信息,多数据库联合分析和多维度图片展示,调用丰富的多组学关联分析工具,深度循环挖掘数据,还可自由上传数据和自定义目的基因分类。
1000+累积影响因子:RNA测序在《Nature》、《Cell》、《Immunity》、《Nature Neuroscience》、《Cell Research》等顶级期刊发表过多篇文章;单细胞转录组文章4月荣登《Genome Biology》,BGISEQ平台定量灵敏准确[4]。
参考文献:
[1] Sayols S, Scherzinger D, Klein H. dupRadar: a Bioconductor package for the assessment of PCR artifacts in RNA-Seq data. BMC Bioinformatics. 2016 Oct 21;17(1):428.
[2] Fu Y, Wu PH, Beane T, et al. Elimination of PCR duplicates in RNA-seq and small RNA-seq using unique molecular identifiers. BMC Genomics. 2018 Jul 13;19(1):531.
[3] Li Q, Zhao X, Zhang W, et al. Reliable Multiplex Sequencing with Rare Index Mis-Assignment on DNB-Based NGS Platform. bioRxiv. 2018: 343137.
[4] Natarajan KN, Miao Z, Jiang M,et al. Comparative analysis of sequencing technologies for single-cell transcriptomics. Genome Biology. 2019 Apr 9;20(1):70.
* 订阅更多关于BGI的资讯请点击这里

