“0”PCR,错误InDel的克星来了!
2019年8月23日-25日,2019全球华人遗传学大会在上海复旦大学盛大举行,华大科技产品总监蔡悦博士受大会邀请,带来了题为《更全面、准确的基因组变异检测新方案》的精彩报告,为基因组变异研究带来了全新的“0”PCR解决方案。
有人说,基因组学可以被划分为两个时代:一个没有PCR,一个有PCR。的确,没有PCR技术,就没有现代分子生物学。PCR(聚合酶链式反应)是一种用于放大扩增特定DNA片段的分子生物学技术,拥有了扩增微量DNA的技术,DNA的测序和探索从此加快了步伐。
高通量测序的文库构建阶段 ,通过PCR将文库模版扩增数千倍,从而达到上机的文库量,以便完成后续对基因组DNA序列的测定。在测序阶段,单个DNA分子的信号难以检测,需要通过PCR扩增使单个DNA分子形成一簇,即cluster,获得足够强度的信号,测序才能够检测到。可见,对于高通量测序,文库构建和测序成簇过程的PCR,是两个关键因素。
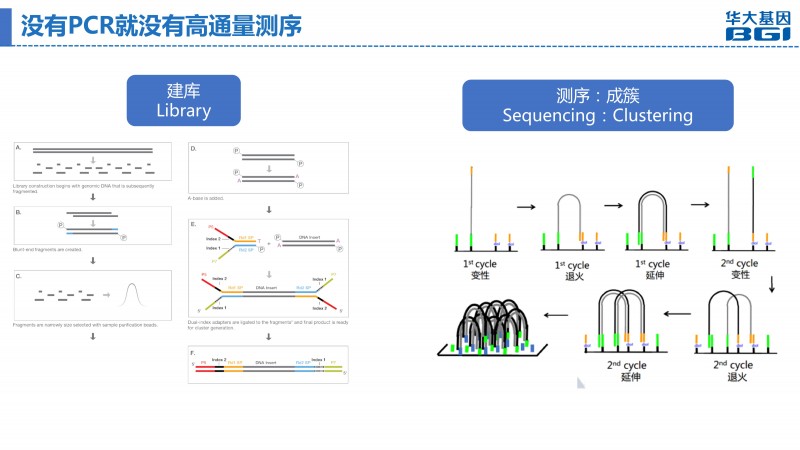
毫不夸张的说,没有PCR,就没有高通量测序。然而,PCR可能带来这样的问题:
错:错误累积[1]
偏:扩增偏向性[2]
多余:Duplicate
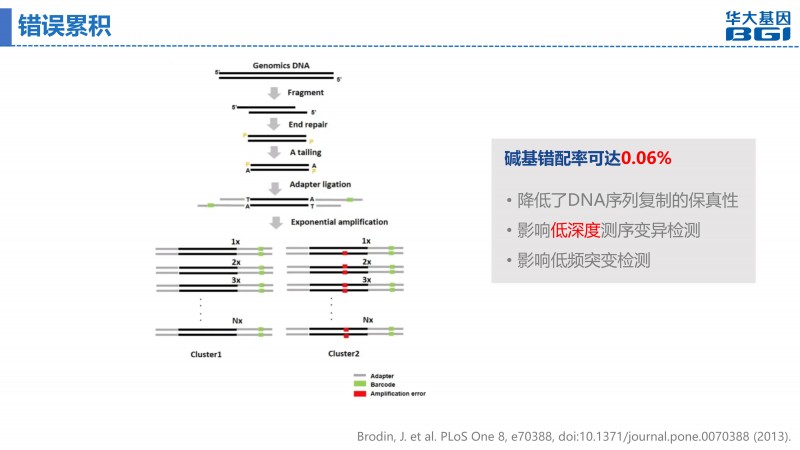
PCR过程中可能引入碱基错配率可达0.06%[3],桥式PCR扩增形成cluster这一过程的错误累积会降低DNA序列复制的保真性,影响低深度测序变异和低频突变检测的精准度和敏感度。
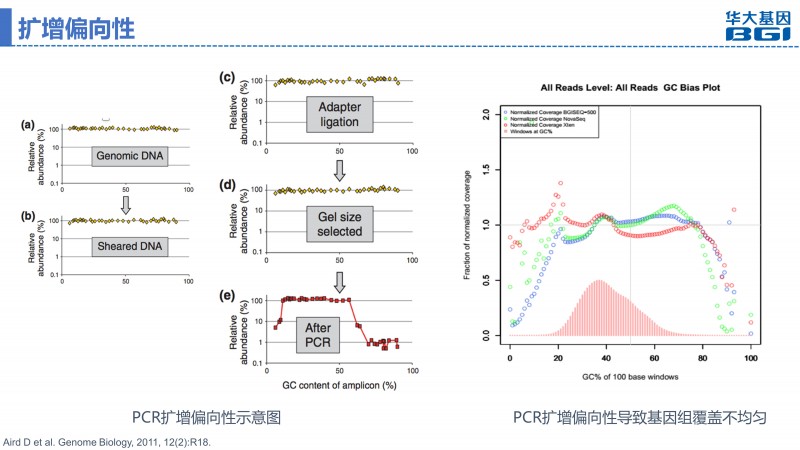
对于核酸模板而言,有的二级结构复杂,有的热稳定性不好,这些因素都会影响PCR扩增效率。因此,并非所有基因组序列都能在PCR扩增文库中同等体现,存在明显的偏向性,会降低基因组的覆盖度。
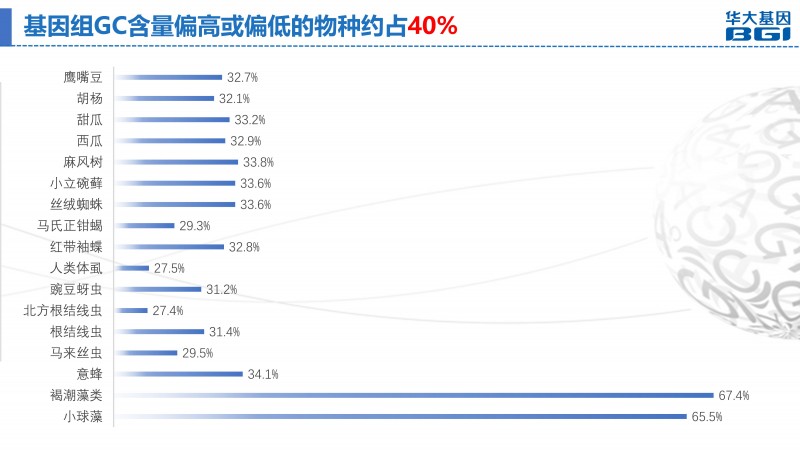
不同物种的GC含量不尽相同,至少有40%的基因组GC含量偏高或者偏低,例如褐潮藻类和小球藻GC含量高达65%以上。对于线虫和马来丝虫,GC含量竟然不到30%。对于这些GC含量偏高或偏低的基因组,想要很好的覆盖基因组存在一定困难。
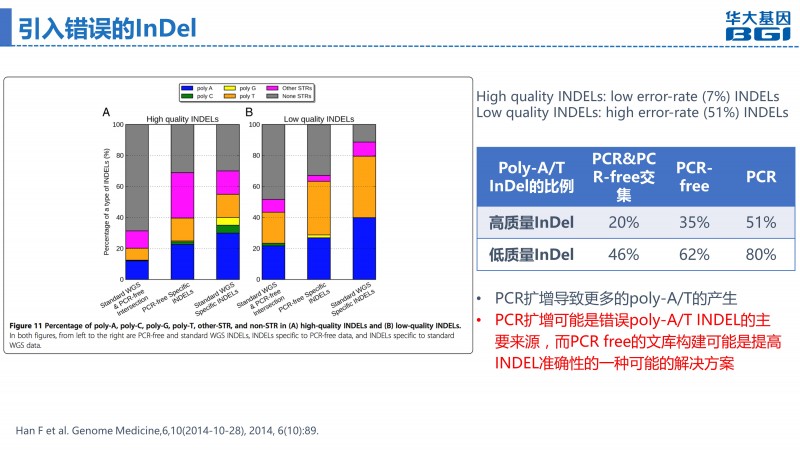
当DNA两条链上的嘌呤嘧啶含量不平衡时会增加缺失发生的几率[4],序列中PolyA结构、 高GC含量的序列会增加复制滑移的几率, 而复制滑移会导致序列发生插入/缺失突变[5],而在这些区域中,PCR扩增会导致明显偏向性,引入大量易出错的InDel。
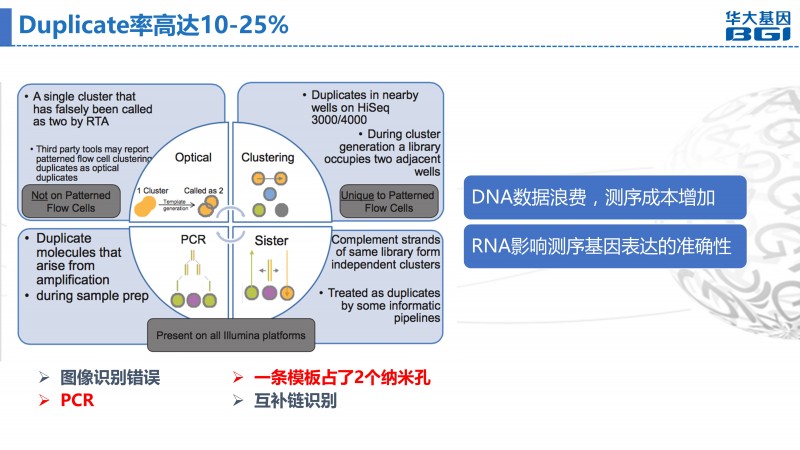
由于建库和测序的PCR扩增,会带来高达10%-25%的duplicate比率,造成DNA数据量浪费,从而增加测序成本。对于RNA来说,因为难以区分是PCR duplicates还是RNA高表达形成的相同的模板,则无法去除duplicates。这将会影响转录组表达量的准确性,尤其是小、中等表达量的转录本的准确性。
由于PCR会带来上述的各种问题,PCR从无到有再到无的过程是高通量测序技术发展的必然趋势。大会上,蔡悦博士提出来PCR-free建库+DNBSEQTM测序技术的“0”PCR测序解决方案,可以更准确、全面的检测基因组变异。
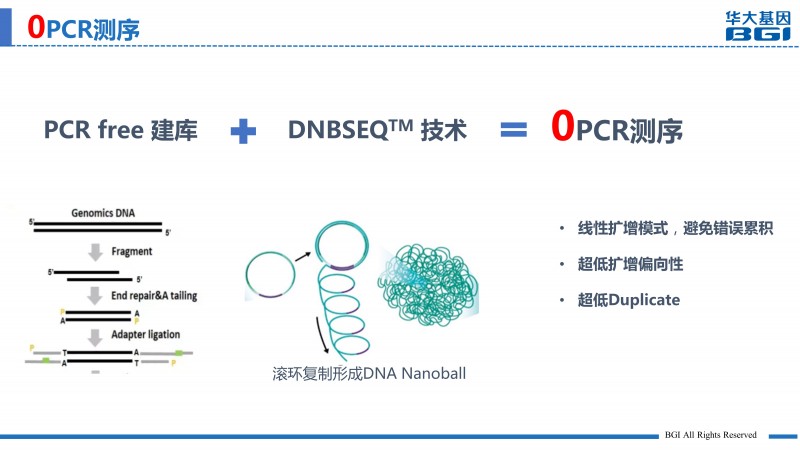
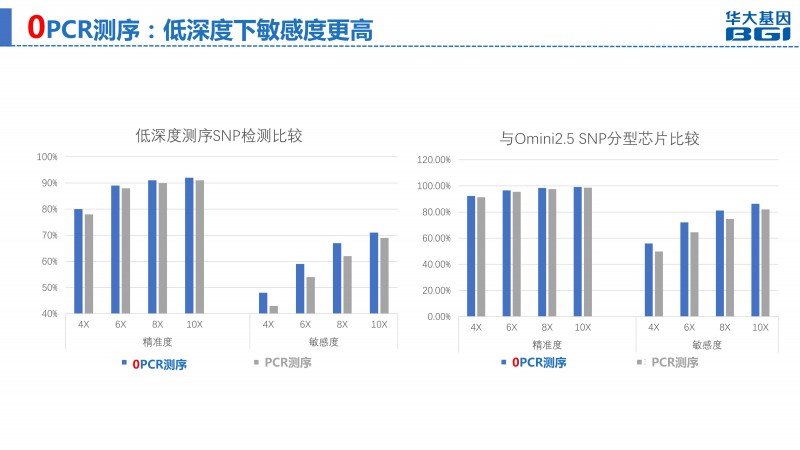
DNBSEQTM技术测序平台,测序过程中采用滚环扩增(Rolling circle amplification,RCA),只以最初的单链环状文库为模板进行线性扩增,不会有PCR过程中错误的累积,真正实现了从建库到测序的“0”PCR,还原最真实的基因组序列。以下是蔡悦博士展示的“0”PCR测序优秀的数据表现:
“0”PCR测序数据展示
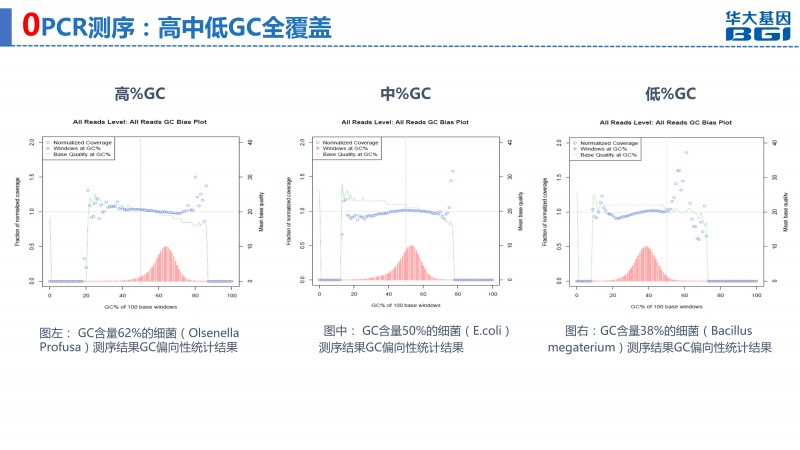
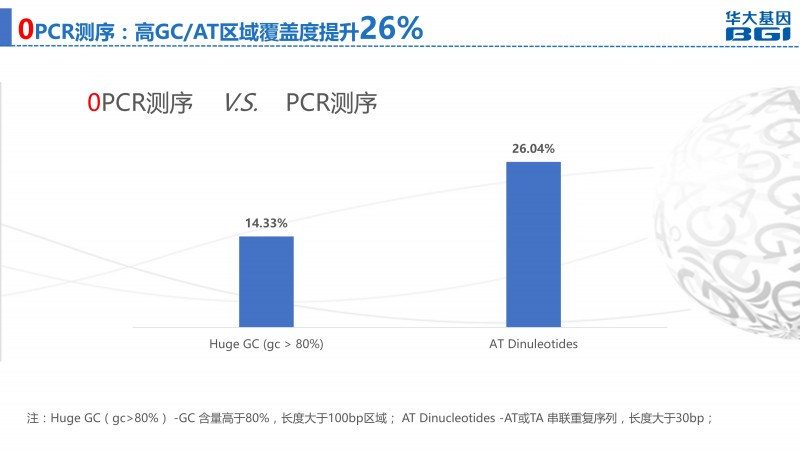
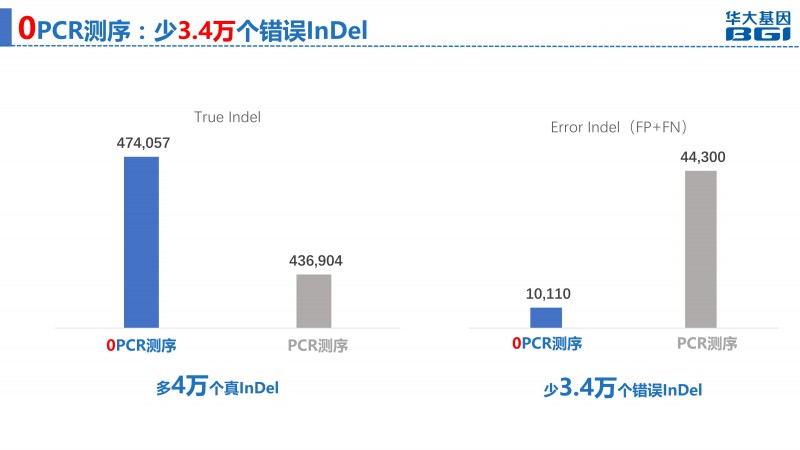
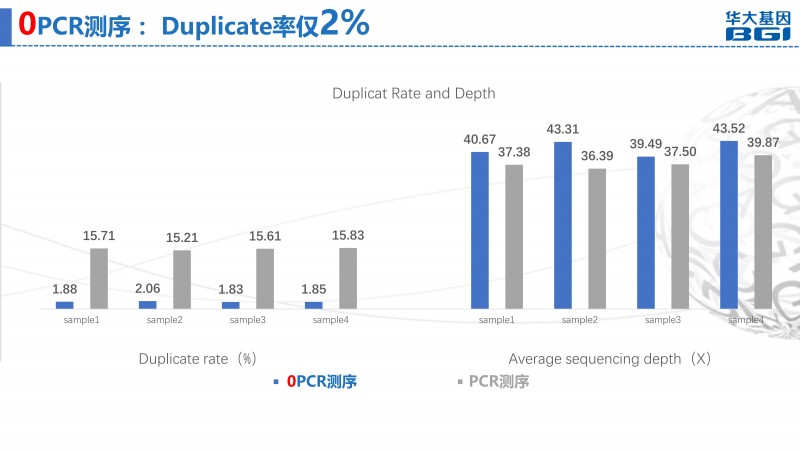
DNBSEQTM“0”PCR测序,带你进入无PCR高通量测序新时代!
参考文献:
【1】Mcinerney P, Adams P, Hadi M Z. Error Rate Comparison during Polymerase Chain Reaction by DNA Polymerase.[J]. Molecular Biology International, 2014, 2014:287430.
【2】Aird D, Ross M G, Chen W S, et al. Analyzing and minimizing PCR amplification bias in Illumina sequencing libraries[J]. Genome Biology, 2011, 12(2):R18.
【3】Johanna B , Mattias M , Charlotte H , et al. PCR-Induced Transitions Are the Major Source of Error in Cleaned Ultra-Deep Pyrosequencing Data[J]. PLoS ONE, 2013, 8(7):e70388-.
【4】 Kondrashov A S, Rogozin I B. Context of deletions and insertions in human coding sequences[J]. Human Mutation, 2004, 23(2):177–185.
【5】 Sjödin P, Bataillon T, Schierup MH (2010) Insertion and deletion processes in recent human history. PloS ONE, 5, e8650.
* 订阅生命科学领域资讯及更多关于BGI的信息请点击这里

