- 首页 > ChIP-Seq
ChIP-Seq
染色质免疫共沉淀(ChIP)是在体内环境中研究蛋白质与DNA相互作用的经典实验方法,广泛应用于组蛋白修饰、特定转录因子的基因调控作用等相关领域。随着新一代测序技术的发展和成熟,染色质免疫沉淀实验与高通量测序的整合——Chromatin
Immunoprecipitation Sequencing (ChIP Sequencing ),可在全基因组范围对蛋白结合位点进行高效而准确的筛选与鉴定,同时也为研究的深入开展打下基础。
采用特异性抗体对目的蛋白进行免疫沉淀后,分离与其结合的基因组DNA片段,再通过高通量测序与数据分析,在全基因组范围内寻找目的蛋白的DNA结合位点,并且可以基于多个样品进行差异比较。
技术流程
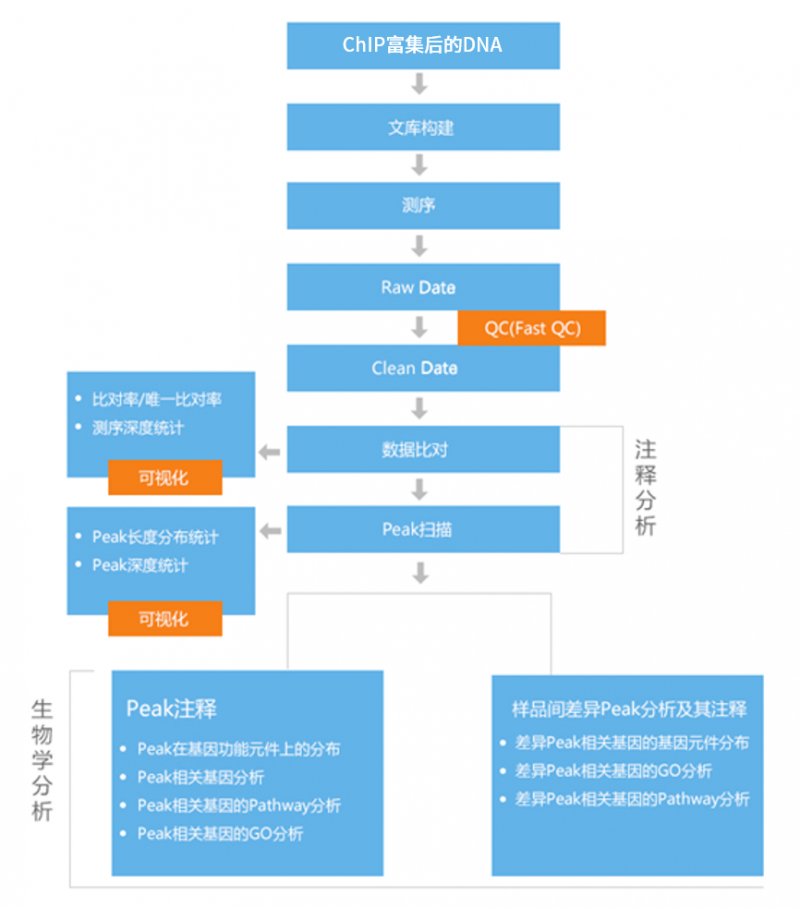
技术优势
周期更短:交付快至25个工作日
低起始量:DNBSEQ平台起始量可低至5ng
准确性高:DNBSEQ与HiSeq平台一致性相关系数高达0.99
关联分析:可定制 ChIP-Seq与RNA-Seq差异表达基因(DEGs)关联分析
研究内容
1) 数据过滤质控;
a) 数据产出统计
b) 质量、碱基比例分布图
2) ChIP 测序序列与参考基因组序列的比对(基于1);
a) 比对结果统计
b) 基因组测序深度累积分布
c) 基因测序深度分布
3) Peak 分析(基于2);
a) Peak扫描
b) Peak长度分布
c) Peak深度分布
4) Peak注释(基于3);
a) Peak在基因功能元件上的分布
b) Peak相关基因分析
c) Peak相关基因的GO功能显著性富集分析
d) Peak相关基因的Pathway功能显著性富集分析
5) 鉴定样品间差异Peak(基于2,3);
对两个样品进行差异分析,确定存在样品间差异修饰的区间。
6) 样品间差异Peak注释(基于5);
a) 差异Peak的基因功能元件分布
b) 差异Peak相关基因分析
c) 差异Peak相关基因的GO功能显著性富集分析
d) 差异Peak相关基因的Pathway功能显著性富集分析
7) 免费提供UCSC genome browser 使用说明(基于2,3)。
定制化信息分析
可结合客户的需求,协商确定定制化信息分析内容(如甲基化与转录组关联分析等)。
案例一:DNBSEQ ChIP-Seq在乳腺癌转移研究中的应用[1]
案例描述: 乳腺癌是人类常见的一种恶性肿瘤,据统计,全球每年新发乳腺癌高达120万人,是女性第一高发的肿瘤,近年来我国的乳腺癌发病率明显增高,而乳腺癌转移是导致患者死亡的主要原因,乳腺癌转移的分子机理目前还不完全清楚,在以往的研究中观察到FOXN3在其他恶性肿瘤中表达失调的现象,但FOXN3在恶性肿瘤(包括乳腺癌)发生中的作用及分子机理仍有待研究,它的功能又需要哪些分子共同作用。
发表单位:北京大学医学部 尚永丰院士
影响因子:12.784
研究技术:(DNBSEQ ChIP-Seq)染色质免疫共沉淀测序、(iRIP-Seq)RNA免疫共沉淀测序等。
研究成果:
揭示了FOXN3-NEAT1-SIN3A阻遏物复合体的存在方式;
全基因组范围内鉴定了FOXN3-NEAT1-SIN3A阻遏物复合体的目标基因;
FOXN3-NEAT1-SIN3A阻遏复合体促进乳腺癌细胞的EMT转化和侵袭;
FOXN3-NEAT1-SIN3A阻遏物复合体促进乳腺癌的转移;
FOXN3和NEAT1在乳腺癌中上调,其高水平与较高的肿瘤分级和较差的存活率相关。
部分结果展示:

图1 A.FOXN3 iRIP-Seq显著富集lncRNA分析结果;B. iRIP-Seq结果qPCR验证结果; C.LncRNA表达芯片检测散点图
案例二:肝细胞中TCF7L2全基因组靶基因调控机制研究[2]
案例描述:TCF7L2在肝脏中代谢通路中具有重要作用,之前的研究发现TCF7L2在成千上万个基因区域有富集,但具体哪一个才是重要的结合位点有待挖掘。
研究思路:

图2 文章研究思路
研究结果:TCF7L2沉默后实时RNA-Seq检测3-96h有406个差异表达基因,进一步结合ChIP-Seq peak相关系数PPS大于10的直接调控Gene有149个,确定TCF7L2参与九大通路调控,其中包括脂质,碳水化合物和氨基酸以及尿素代谢途径等。

图3 TCF7L2 peak proximity scores与DEGs表达差异值趋势图

图4 不同时期peak proximity scores与DEGs关系分布图
参考文献
[1]Yongfeng Shang,Yu Zhang,et al.The FOXN3-NEAT1-SIN3A repressor complex promotes progression of hormonally responsive breast cancer. J Clin Invest. 2017 Aug.
[2]Luke N., et al. (2014) The mechanisms of genome-wide target gene regulation by TCF7L2 in liver cells. Nucleic Acids Research, 2014, Vol. 42, No. 22 13647.
DNBSEQ ChIP-Seq平台及建库技术简介
1. 平台介绍
DNBSEQ平台已经广泛用于DNA、RNA测序,基于此平台不断有高分文章产出。DNBSEQ测序平台有五大关键的技术:DNB、Pattern array、cPAS、MDA-PE、sCMOS,保证了该平台测序的准确性。
与其他二代测序技术相比较,DNB测序技术具有以下几个优势:
(1)DNB通过增加待测DNA的拷贝数而增强了信号强度,从而提高测序准确度;
(2)不同于PCR指数扩增,滚环扩增技术的扩增错误不会累积;
(3)DNB与芯片上活化位点的大小相同,每个位点只固定一个DNB,保证信号点之间不产生相互干扰;
(4)阵列化测序芯片和DNB测序技术的结合,使得成像系统像素和测序芯片的面积得到最大化利用。
2. 建库技术

图1 DNBSEQ平台ChIP-Seq建库测序流程图
DNBSEQ ChIP-Seq数据展示
1. 与HiSeq测序平台一致性相关系数高达0.99

图2 DNBSEQ平台与HiSeq平台ChIP-Seq结果一致性评估
2. 测序重复性高达0.98


图3 DNBSEQ平台ChIP-Seq测序重复性评估 图4 HiSeq平台ChIP-Seq测序重复性评估
3. 全面的信息分析,蛋白位点无处可逃

图5 peak在基因功能元件上的分布分析 图6 peak相关基因的GO功能显著性富集分析
样品要求
样品类型:IP富集DNA样品,强烈建议同时送对应的Input DNA;
总量:≥ 10 ng ChIPed DNA(人类样本可以低至5ng);
浓度:≥ 1 ng/μL;
纯度:OD260/280= 1.8 -2.0;
DNA片段大小:分布在100~500 bp范围,且主带明显。请提供DNA打断后的检测胶图以确定DNA片段大小是否符合要求,并请附加一份详细的样品信息单并提供ChIP后的q-PCR验证结果;
执行周期
46个样本以下,25个工作日(DNBSEQ平台)
推荐数据
推荐数据量: 20M或40M clean reads。
Q1:DNBSEQ平台的测序原理是什么?
DNBSEQ采用优化的联合探针锚定聚合技术(cPAS)和改进的DNA纳米球(DNB)核心测序技术,是行业领先的高通量测序平台之一。具体而言,首先DNA分子锚和荧光探针在纳米球上进行聚合,随后高分辨率成像系统对光信号进行采集,光信号经过数字化处理后即可获得待测序列。其中,DNB通过线性扩增增强信号,降低单拷贝的错误率。此外,DNB大小与芯片上活性位点的大小相匹配,每个位点结合一个DNA纳米球,在保证测序精度的情况下提高了测序芯片的利用效率。
Q2:DNBSEQ ChIP-Seq产品优势是什么?
a)
准确性高:高深度Peaks与HiSeq平台一致性高达到100%
b) 起始量低:样品起始量低至5ng
c) 周期更短:交付快至25个工作日
d) 性价比高:相同的测序质量,价格更优惠
e) 关联分析:ChIP-Seq与RNA-Seq差异表达基因(DEGs)关联分析
Q3:对样本起始量有没有要求?
建库起始量为5ng,建议ChIP-ed DNA≥ 20ng,低于5ng也可以做,之前有做过无浓度样本成功的案例,但这类项目只能客户自己承担建库风险。
Q4:测序策略是什么?
SE50
Q5: ChIP-Seq的对照中input和IgG有什么不同吗?
Input对照:在进行免疫沉淀前,需要取一部分断裂后的染色质做Input对照。Input是断裂后的基因组DNA,不加抗体做富集,但是需要与沉淀后的样品DNA一起经过逆转交联,DNA纯化,以及最后的PCR或其他方法检测。Input对照不仅可以验证染色质断裂的效果,还可以根据Input中的靶序列的含量以及染色质沉淀中的靶序列的含量,按照取样比例换算出ChIP的效率,所以Input对照是ChIP实验必不可少的步骤。
阴性对照:用普通的IgG做为抗体(目的蛋白抗体宿主的IgG或血清)。理论上不会ChIP下来任何DNA片段,因此作为阴性对照,但是由于非特异结合,或者实验过程中,没发生结合的DNA清除不完全,可能也会出现条带,如果非常明显,那就证明实验过程有待改进。
Q6:推荐多少数据量?能否接不同数据量的项目?
DNBSEQ ChIP-Seq推荐20M clean reads/样本,其他类型数据量也可提供。
Q7:N-ChIP和X-ChIP的区别是什么?
N-ChIP基于核酸内切酶MNase酶切,切割核小体且作用温和,较适用于组蛋白修饰研究;X-ChIP基于甲醛固定,超声打断,适用于大多数蛋白-DNA相互作用研究。
Q8:样品制备过程是否需要PCR扩增?PCR扩增后是否会影响最后的结果?
基于第二代测序平台的ChIP-Seq在样品制备过程中需要进行PCR,但是只需要非常少的反应次数,由于PCR引起的偏向性非常小。另外,在测序结果出来后,针对reads的比对结果,通过信息分析手段可以去除duplication的影响。我们的所有信息分析结果都是在去除duplication之后进行生物学意义挖掘的。根据我们的经验,如果您送的样品是直接ChIP富集的DNA,在我们的样品制备后出现duplication的reads比例不会超过全部reads产量的5%。如果您送的样品在ChIP富集后有做过PCR扩增,那么这一步的PCR对结果的duplication影响非常严重。
Q9:哪些因素会影响ChIP-Seq的结果?
抗体的质量与特异性、需要富集的目标区域在基因组上的比例、ChIP的实验操作、DNA片段长度范围等都会影响ChIP-Seq的结果。

