- 首页 > DNBelab C系列高通量单细胞RNA-Seq
DNBelab C系列高通量单细胞RNA-Seq
DNBelab C系列平台基于液滴微流控策略,可以实现高效细胞捕获,完成高效的mRNA反转录和扩增后,结合华大DNBSEQ™高通量测序平台和Dr. Tom单细胞全交互式数据挖掘交付系统,进行高性价比单细胞转录组文库测序和数据分析挖掘,可广泛应用于研究组织异质性、胚胎发育、克隆进化、疾病机理和免疫微环境等。

图1 DNBelab C4仪器
产品优势
1. 稀有细胞高效捕:高效捕获稀有类型细胞,实现新突破;
2. 多种样本无挑剔:除常规活细胞悬液外,珍稀液氮速冻样本可实现细胞核水平研究;
3. 测序数据高保真:搭配DNBSEQ™测序系统,不怕单个细胞数据弄混;
4. 自主分析不求人:Dr. Tom交互式单细胞数据挖掘系统支持;
5. 服务模式一站达:流式分选服务服务助力目的细胞分选,一站式实现从组织到细胞数据获取及细胞群验证。
产品性能
- 高灵敏度细胞分选:高效捕获稀有细胞类型
将两种不同状态的人多功能干细胞(PPSCs以及NPSCs)以99:1的比例混合后,进行DNBelab C系列平台单细胞RNA文库制备和测序,最后鉴定到NPSCs细胞比例为1.3%,表明该平台具备可靠的罕见细胞捕获能力。
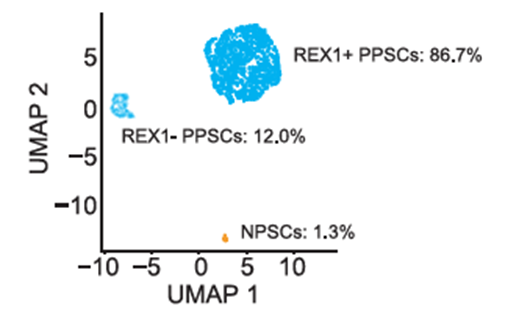
图2 各类细胞分选检出比例
- 样本无挑剔:除常规单细胞悬液样本之外,珍稀液氮速冻样本可实现细胞核水平研究
采用液氮速冻的肺组织样本,提取细胞核,进行DNBelab C系列平台单细胞分选建库及测序,共获得12,822个细胞核,每个细胞核检测基因数中位数1,000以上,助力珍稀样本实现单细胞(核)水平研究。
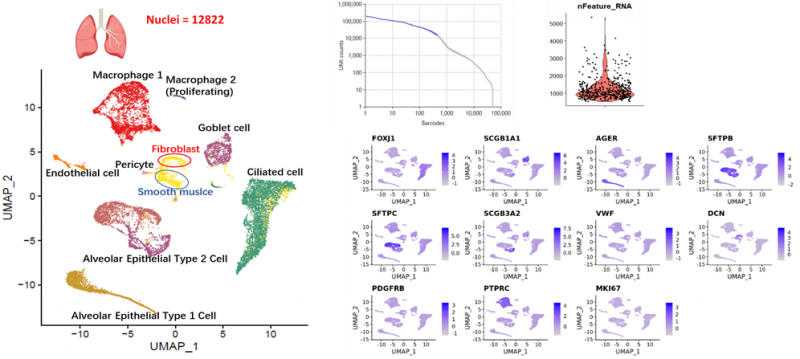
图3 液氮速冻肺组织样本提取细胞核分选结果展示
- 有效细胞分群:与相同类型样本在其他平台已发表分群结果重合度高
DNBelab C系列平台对人外周血PBMC样本进行高效细胞分选和转录组建库测序,所得单细胞数据UMAP降维分群后,与2020年Nature Biotechnology已发表文章中人PBMC细胞分类及注释结果均一致[1]。
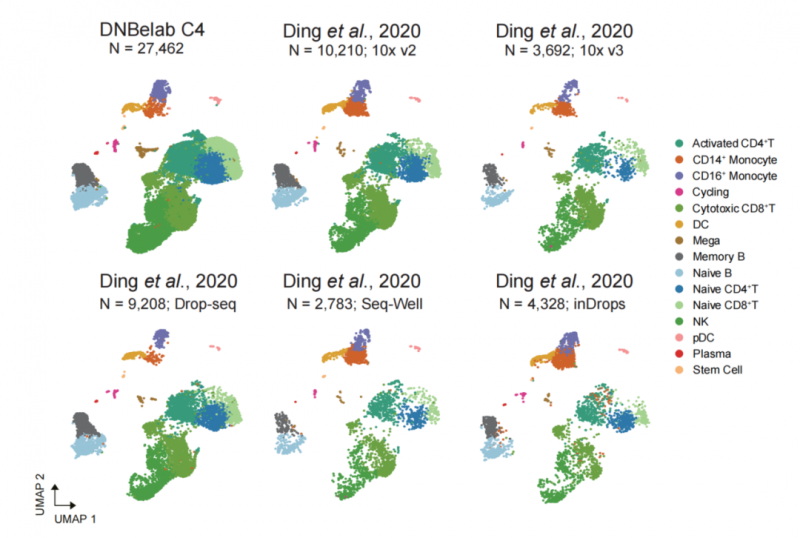
图4 DNBelab C系列平台的细胞分群对比
- 发表多篇高分成果
DNBelab C系列单细胞分选平台已助力发表文章28篇,平均影响因子多达17+。

图5 DNBelab C系列平台发表文章列表
实验流程
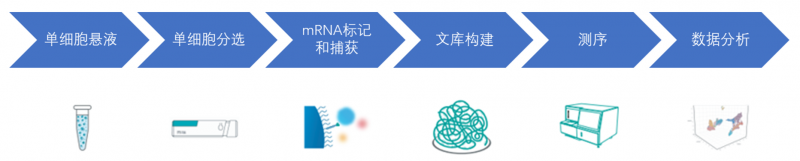
图6 DNBelab C系列平台单细胞技术实验全流程
产品类型
标准通量——预计产出5,000-10,000个单细胞/样本
*以上版本均包含建库、测序和Dr. Tom标准分析
测序策略
推荐DNBSEQ™平台测序:
测序策略:PE100
测序深度:推荐平均50k reads/cell
生信分析内容
- 分析内容
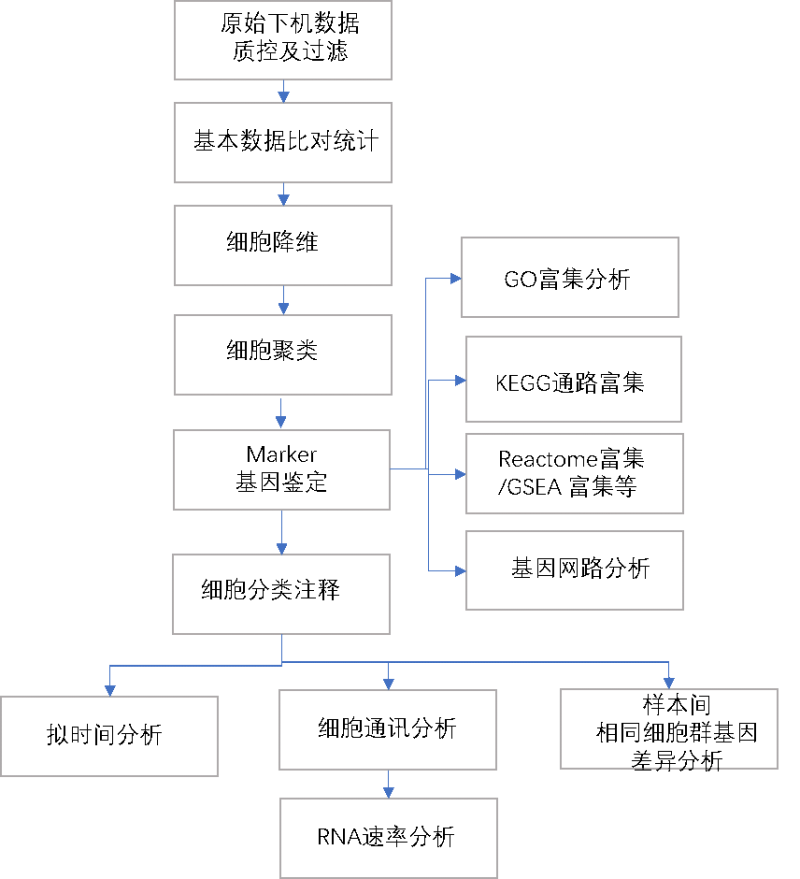
图7 信息分析思路图
- 分析交付-采用Dr. Tom 系统
https://biosys.bgi.com/#/report/login
参考文献:
[1] Ding Jiarui,Adiconis Xian,Simmons Sean K et al. Systematic comparison of single-cell and single-nucleus RNA-sequencing methods.[J] .Nat Biotechnol, 2020, 38: 737-746.
案例一:全球首个非人灵长类动物(猕猴)全身器官细胞图谱[1]

文章题目:
Cell transcriptomic atlas of the non-human primate Macaca fascicularis
发表时间:2022年4月13日
发表期刊:Nature(IF= 69.504)
方案设计:采集成年猕猴的45个组织,基于华大自主DNBelab C系列平台进行高通量单细胞核/单细胞转录组(sn/scRNA-seq)测序,共获得约114万个高质量的单细胞/单细胞核数据。
研究结论:
该研究详细分析了共有细胞组织特异性分子,成年潜在干细胞类型,病毒易感细胞类型数据库,人类表型和疾病变异的细胞定位图谱。与此同时,研究人员搭建了非人灵长类动物百万单细胞交互式资源网站(https://db.cngb.org/nhpca/),为生物医学的发展提供一个基础性的资源和工具,为疾病诊疗、靶向药物开发提供助力,为人类更好地探究生命的进化提供可能。
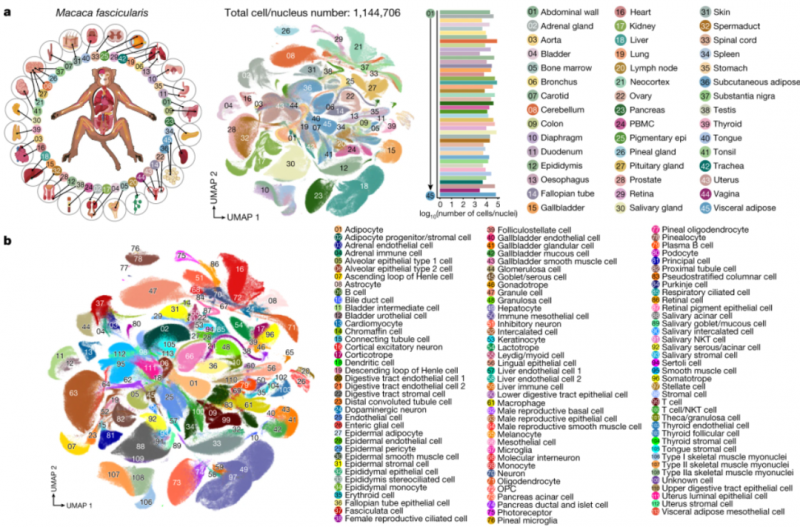
图1 猕猴全身器官细胞图谱
案例二:新冠患者外周血免疫细胞的整体免疫反应特征[2]

文章题目:
Single-Cell Sequencing of Peripheral Mononuclear Cells Reveals Distinct Immune Response Landscapes of COVID-19 and Influenza Patients
发表时间:2020年7月19日
发表期刊:Immunity(IF=43.474)
发表单位:中国疾病预防控制中心病毒病预防控制所、深圳华大生命科学研究院、解放军总医院第五医学中心、北京地坛医院和深圳国家基因库
方案设计:10 名受试者,包括 3 名健康供体、2 名甲型流感病毒(IAV)和5 名 COVID-19感染患者的外周血样本,进行DNBelab C4 高通量单细胞转录组测序,共分析46,022 个细胞
技术路线:

研究结论:
相较于正常人,新冠肺炎患者和甲流患者的外周血中负责产生抗体的浆细胞数目都显著增加。而XAF1-,TNF-和FAS-等凋亡通路相关基因在新冠患者外周血中特异性上调,预示了由这些基因诱导的T细胞凋亡可能是导致患者外周血中T细胞减少的原因之一。进一步的分析显示,与甲流患者相比,新冠患者免疫反应激活的信号通路存在显著性差异:新冠患者以STAT1和IRF3上调为主,而甲流患者则是以STAT3和NFκB为主。文章中系统描绘了新冠患者外周血免疫细胞的整体免疫反应特征,阐述了新冠和甲流感染免疫特征的异同点,为理解新冠的发病机制、指导临床诊治和疫苗的研发及应用提供了重要的参考。
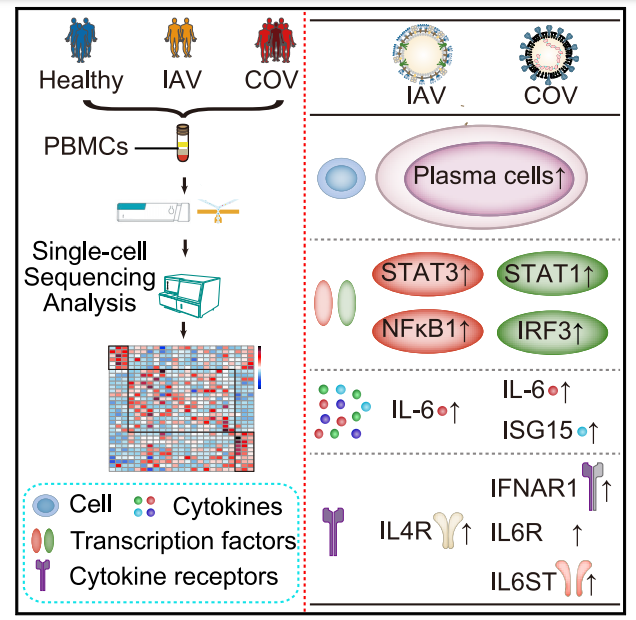
图2 新冠患者外周血免疫细胞的整体免疫反应特征及结果概览
案例三:首个蚂蚁全品级大脑单细胞图谱[3]

文章题目:
A single-cell transcriptomic atlas tracking the neural basis of division of labor in an ant superorganism
发表时间:2022年6月16日
发表期刊:Nature Ecology & Evolution(IF= 19.1)
方案设计:选用社会性昆虫研究的模式物种——法老蚁作为研究对象,采用DNBelab C系列单细胞分选平台,对来自4种不同品级(蚁后、处女繁殖蚁、工蚁和雄蚁)的法老蚁的17个样本 [处女繁殖蚁(4)、蚁后(4)、雄蚁(4)和工蚁(5)] 的大脑,进行高通量单细胞核转录组研究,共获得206,367个单细胞核。
技术路线:
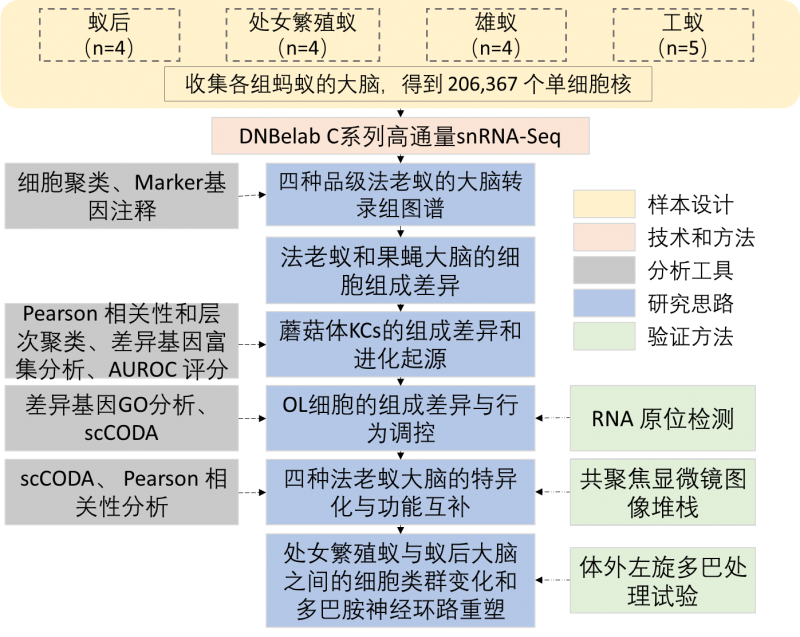
研究结论:
与果蝇不同,蘑菇体KCs在法老蚁中大量存在,并且表现出高度的多样性,其中大多数亚型在工蚁大脑中富集,而负责处理视觉信息的视叶细胞在工蚁大脑里的含量则很低。雄蚁与工蚁脑细胞组成的趋势相反,处女繁殖蚁和蚁后的大脑细胞类型则相对“正常”。研究团队还将处女繁殖蚁与成熟蚁后的大脑进行比较,发现视叶细胞的含量在蚁后大脑中明显降低,这与蚁后长期适应黑暗的巢内环境相关;而多巴胺细胞的含量则在蚁后脑中显著增加,发挥促性腺功能。永久品级和性别分化导致了蚂蚁大脑细胞类群和神经回路的特异性变化。
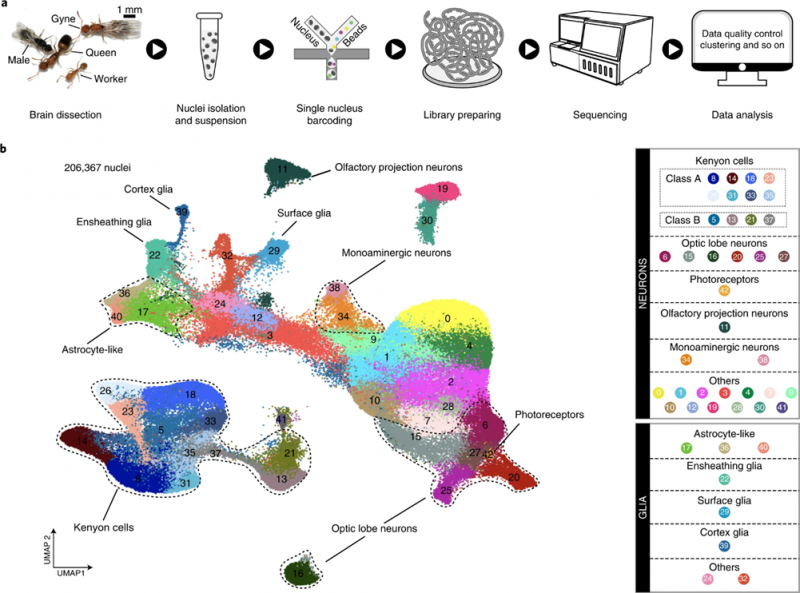
图3 蚂蚁大脑单细胞转录组实验设计和细胞图谱组成
案例四:首个家猪多组织器官的单细胞转录组图谱[4]

文章题目:
Endothelial cell heterogeneity and microglia regulons revealed by a pig cell landscape at single-cell level
发表时间:2022年6月24日
发表期刊:Nature Communications(IF= 17.694)
方案设计:共收集20 种猪组织/器官,其中9个组织通过高通量单细胞转录组测序 (scRNA-seq) 分析,包括内脏和皮下脂肪、脾脏、肠、肝脏、肺、外周血单个核细胞 (PBMC)、全脑和视网膜;15个组织通过高通量单核转录组测序 (snRNA-seq) 分析,包括 9 个脑区(后区、小脑、穹窿下器官、终板血管器官、额叶、下丘脑、枕叶、顶叶和颞叶)、视网膜、肾脏、心脏、脾脏、肝脏和肺;4种猪组织(肝脏、视网膜、肺、脾)同时采用 scRNA-seq 和 snRNA-seq 分析;最终获得133,492个细胞和 89,034 个细胞核转录组数据。
技术路线:
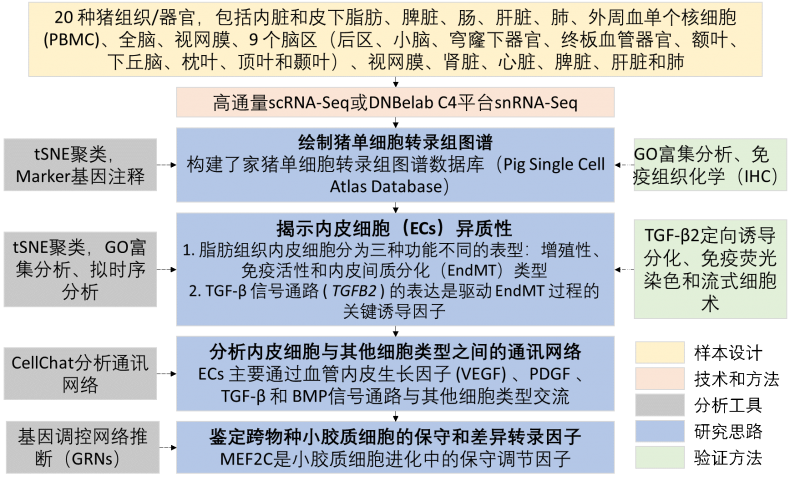
研究结论:
该研究应用单细胞转录组测序首次构建家猪多组织和器官的细胞图谱,证实猪内皮细胞存在着高度的细胞和功能异质性;接着发现和验证了脂肪组织中存在内皮间质转分化细胞亚型(EndMT),以及证明TGF-β2信号通路在EndMT转分化中的关键作用。该研究进一步揭示了脑小胶质细胞遗传保守转录调控网络。以上发现为利用家猪研究人心血管疾病和改善异种器官移植免疫排斥反应提供关键科研资源和科学基础,同时为跨物种的小胶质细胞进化提供了保守和差异转录因子的宝贵资源,对人脑疾病中的小胶质细胞进化模块研究具有重要意义。
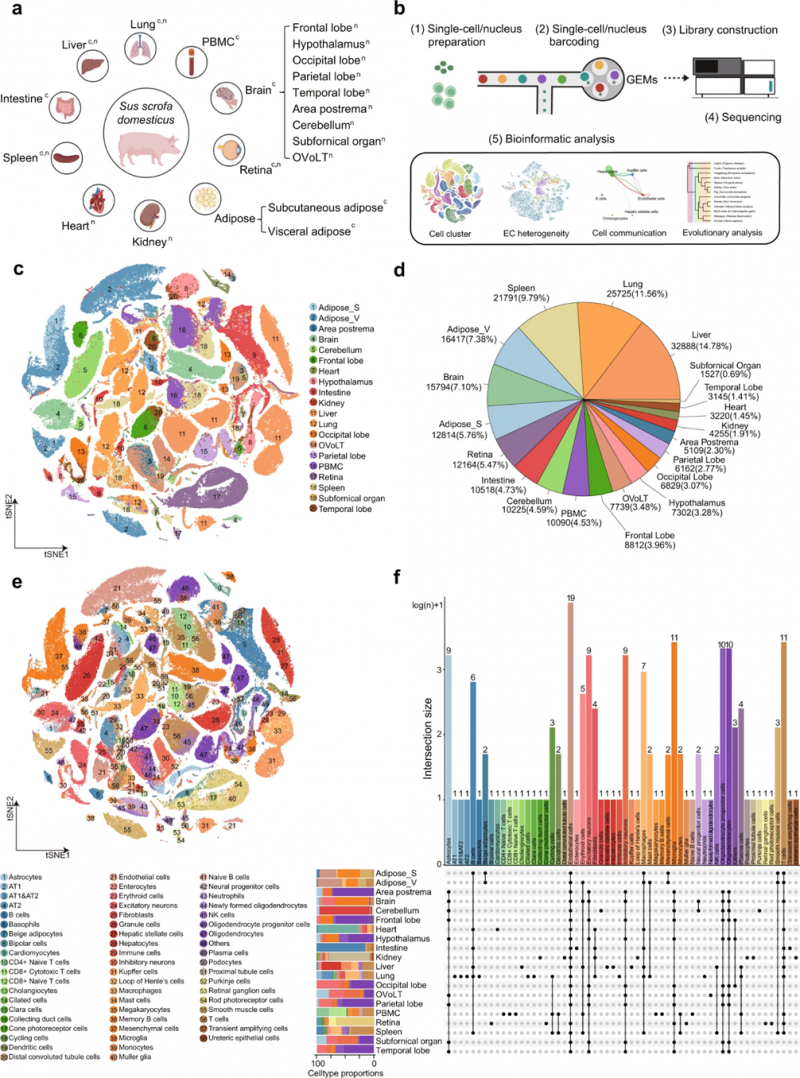
图4 20种猪组织的单细胞转录组图谱
案例五:多区域单细胞测序揭示结直肠癌免疫微环境转录组学特征[5]

文章题目:
Multiregion Single-Cell Sequencing Reveals the Transcriptional Landscape of the Immune Microenvironment of Colorectal Cancer
发表时间:2021年1月1日
发表期刊:Clinical and Translational Medicine(IF=8.554)
发表单位:广东省人民医院姚学清教授团队
方案设计:收集18位原发性和肝转移CRC患者的肿瘤样本,利用流式技术分选出免疫细胞和非免疫细胞,分别采用两种单细胞RNA测序技术(Smart-seq2 和 DNBelab C4 技术),测序细胞总数达到15,115个
研究结论:
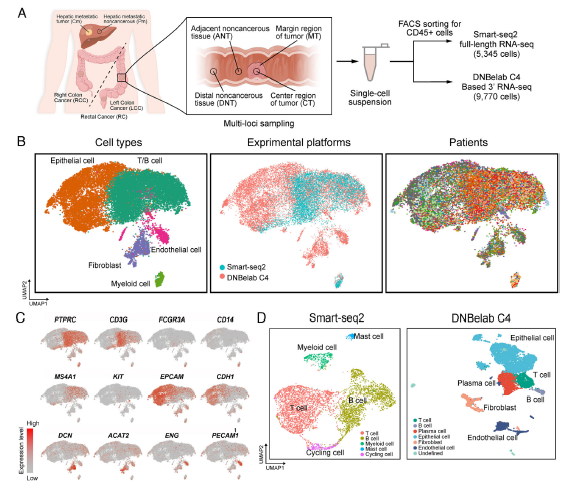
图5 结直肠腺癌(CRC)肿瘤和非肿瘤样本的免疫图谱
与非肿瘤组织相比,肿瘤组织中T细胞和B细胞在亚群和功能状态上都发生显著变化:其中早期CRC肿瘤B细胞为具有抗肿瘤能力的pre-B样细胞,而晚期CRC患者的B细胞倾向于发育成浆细胞。另外,还发现IgA+IGLC2+浆细胞与CRC预后不良有关,说明B细胞和髓细胞之间信号通路往来十分密切。而且CCL8+ 循环B细胞和CCR5+ T细胞之间的相互作用在晚期CRC患者发挥着潜在的抗肿瘤作用。本项研究对CRC的免疫浸润现象进行了十分深入的探索,为未来CRC的免疫治疗研究提供了新的思路。
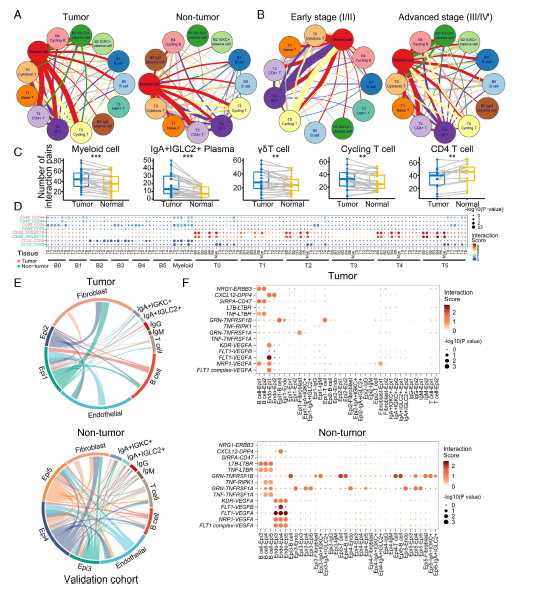
图3 结直肠腺癌(CRC)肿瘤微环境中的细胞-细胞通讯
参考文献:
[1] Han L, Wei X, Liu C, et al. Cell transcriptomic atlas of the non-human primate Macaca fascicularis. Nature. 2022;604(7907):723-731. doi:10.1038/s41586-022-04587-3
[2] Zhu L, Yang P, Zhao Y, et al. Single-Cell Sequencing of Peripheral Mononuclear Cells Reveals Distinct Immune Response Landscapes of COVID-19 and Influenza Patients. Immunity. 2020;53(3):685-696.e3. doi:10.1016/j.immuni.2020.07.009
[3] Li Q, Wang M, Zhang P, et al. A single-cell transcriptomic atlas tracking the neural basis of division of labour in an ant superorganism. Nat Ecol Evol. 2022;6(8):1191-1204. doi:10.1038/s41559-022-01784-1
[4] Wang F, Ding P, Liang X, et al. Endothelial cell heterogeneity and microglia regulons revealed by a pig cell landscape at single-cell level [published correction appears in Nat Commun. 2022 Nov 8;13(1):6748]. Nat Commun. 2022;13(1):3620. Published 2022 Jun 24. doi:10.1038/s41467-022-31388-z
[5] Wang W, Zhong Y, Zhuang Z, et al. Multiregion single-cell sequencing reveals the transcriptional landscape of the immune microenvironment of colorectal cancer. Clin Transl Med. 2021;11(1):e253. doi:10.1002/ctm2.253
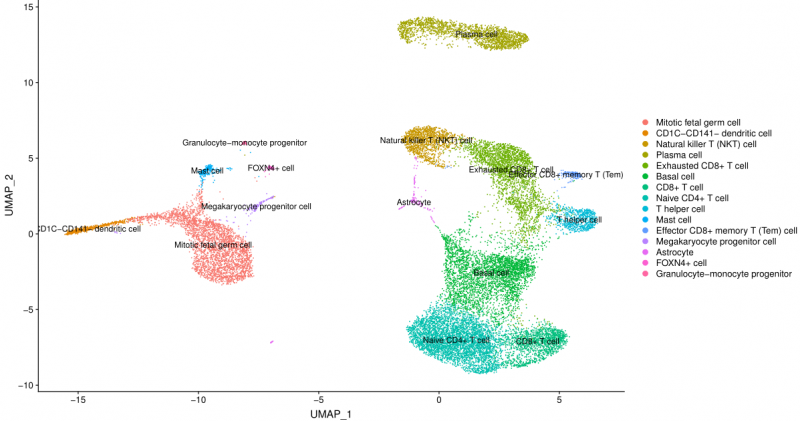
图1 细胞分类注释图
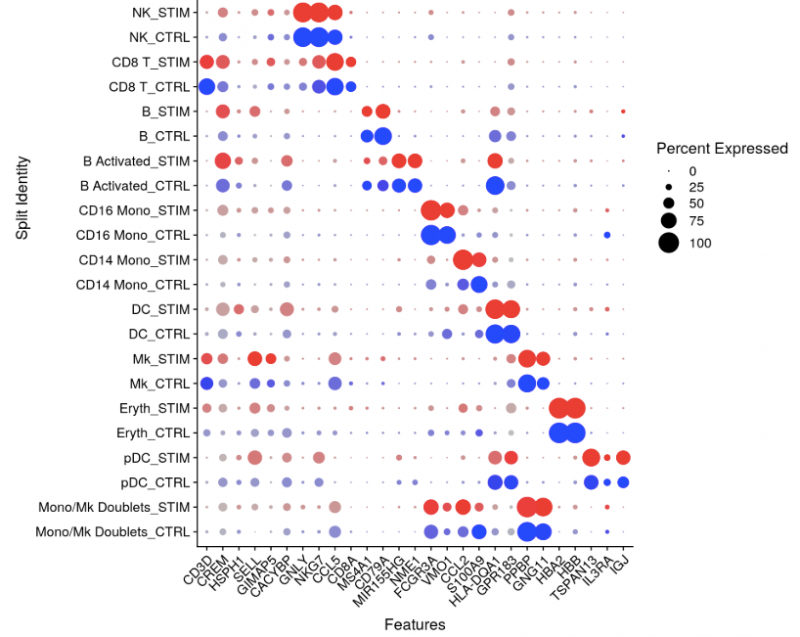
图2 cluster特异marker基因点图
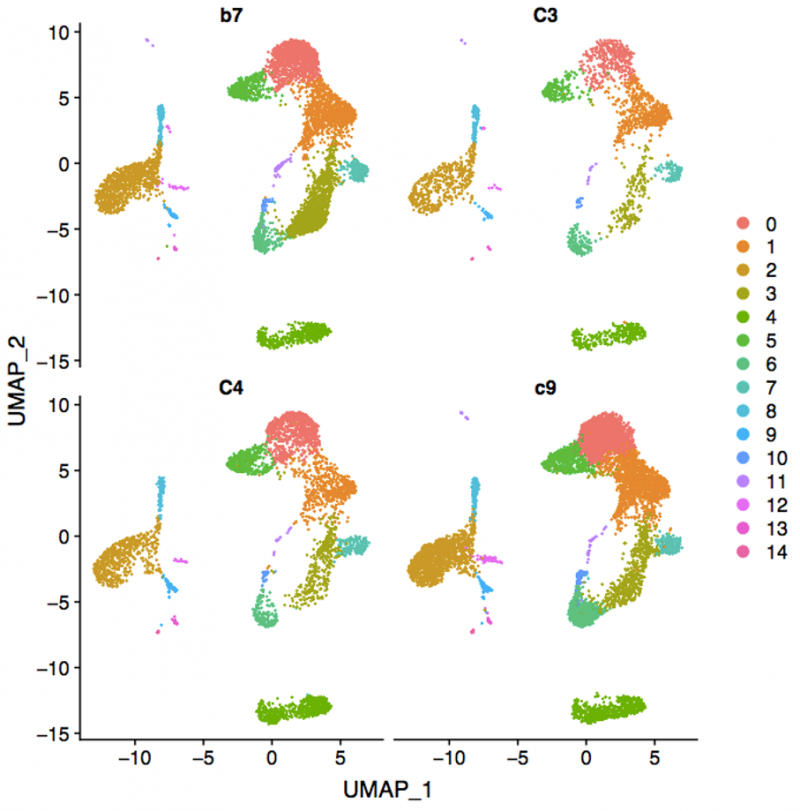
图3 各样本细胞类型聚类对比图
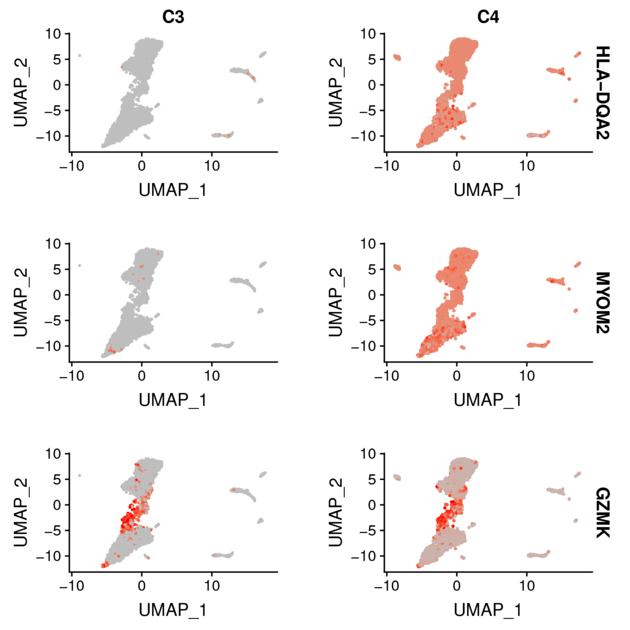
图4 样品间相同细胞类型差异基因点阵热图
|
样本 要求 |
类型 |
其他组织(细胞):人-肺、肾脏、肿瘤类、淋巴结; 小鼠-肺、脾脏、肾脏、淋巴结、皮肤 脑组织(细胞核):人、 小鼠 |
|
细胞:PBMC、细胞系 |
||
|
细胞状态 |
细胞直径<=40μm;细胞活性>80%;细胞浓度 > 1000细胞/μL左右(950~1050个/μL),推荐送样 ≥ 细胞总量 1*105个 |
|
|
细胞悬液背景干净,结团率需控制在10%以下,无碎片及杂质,不含Ca2+和Mg2+ |
交付周期
从样品复苏后,镜检符合上机要求、预付款到位开始(不包括由于样品等问题停滞时间以及试剂订购周期),细胞类型及生物信息分析整体完成周期视不同的类型而有所变化。
- 样品数≤8个,40个自然日(含建库、测序、标准信息分析)。
- 样品数≥9个,请单独咨询周期。
Q1. 通过DNBelab C系列平台进行单细胞RNA-Seq的组织样品,是否可以提供前期处理的方法?
答: 目前单细胞送样建议手册上有提供通用的组织消化方法,如下可供参考。
需要注意,不同组织存在组织及细胞的特异性,使用通用组织消化方法的效率会因组织类型差异,存在消化效率的差异。因此,建议客户自行使用组织特异性的针对性消化方法进行消化。
Q2. DNBelab C系列平台分离的单细胞会有细胞大小的限制吗?
答: 细胞大小会有一定的限制,但限制条件相对固体芯片没有那么严格。同时,生产实验过程中,为了避免细胞出现结团情况,在进行细胞分离处理前,会做一个粗略的过筛处理。建议细胞大小<40um。
Q3. 请问DNBelab C系列平台单细胞RNA-Seq可以进行可变剪切等结构变化分析吗?
答: DNBelab C系列平台单细胞RNA-Seq主要定位为基因表达定量分析为主,数据不建议进行可变剪切等基因结构变化分析。如需研究结构变异信息,可进行单管单细胞RNA测序。
Q4. DNBelab C系列平台单细胞RNA-Seq采用的3’端扩增技术与Smart-seq2的扩增技术有什么区别?
答: Smart-seq2扩增技术针对是的3’~5’端全部的mRNA;3’端扩增技术主要是扩增3’端,是否能延伸至5’端,取决于酶的活性等系列实验因素决定。因此,扩增产物的长度上会有一定的区别和差异。
Q5. 细胞物种没有相应的参考基因组,可以做这个产品吗?
答: 产品分析需要基于一个良好注释和组装的参考序列。基因注释不完善或只注释到转录本,没有注释到基因等的不完善参考序列,会出现占用内存过多,即使对转录本进行去冗余及重新构建,跑出的结果可能会不太好,需要客户明确其风险因素。
Q6. DNBelab C系列平台产生的Raw data是采用什么平台分析?
答:DNBelab C系列平台有配套的数据分析网站DNBelab_C,具体网址为:https://github.com/MGI-tech-bioinformatics/DNBelab_C_Series_scRNA-analysis-software

