- 首页 > 脂质组学
脂质组学
脂质组学是对生物体内的脂质进行系统分析的一门新兴学科, 是代谢组学的重要分支。基于液质联用技术(LC-MS),无偏向性、尽可能多地检测细胞、组织、器官或体液等生物样本内所有的脂质分子,对实验组和对照组进行对比分析,通过统计分析筛选差异脂质分子, 进而寻找脂质代谢变化与生理病理变化的相对关系,揭示脂质在各种生命活动中的作用机制。
技术优势
1. 脂质分子鉴定准确度高
比对二级质谱图鉴定,鉴定数1000左右,进行脂质大类、亚类、种属、分子多层次分析
2. 超高分辨率质谱平台
使用 Orbitrap 系列谱仪进行检测,仪器分辨率高、质量精度高、稳定性好
3. 规范化平台严格质控
全流程标准操作程序(SOP)指导,同位素内标与QC双重质控
4. 大样本项目经验丰富
高通量自动化样本制备系统进行代谢物提取,实时监控仪器检测过程
5. 分析创新
创新性引入相关性差异分析,通过多维信息整合,稳健算法直击核心功能,精准锁定关键机制。
产品应用
1. 脂质的鉴定与功能研究
2. 脂质生物标志物发掘
3. 脂质代谢途径和网络研究
技术路线

技术参数
1. 仪器平台
LC-MS/MS:液相-Waters ACQUITY UPLC,质谱-Thermo Q Exactive / Q Exactive HF / Q Exactive HF-X
2. 分析软件
LipidSearch(Thermo, USA),metaX (华大自主开发的代谢组学分析软件包)
3. 数据库
LipidSearch配套数据库,脂质组数据库,包含一级质谱和二级质谱信息,覆盖8大类脂质、300种脂质亚类,包含超过170万个脂质离子及其预测碎片离子的信息
项目周期
30-60个自然日
案例一: 首个中国人群糖尿病非靶向血浆脂质组研究(华大合作发表)[1]
研究背景:
血脂异常和II型糖尿病(T2D)之间的联系已经被广泛报道,但II型糖尿病发展过程中的脂质全谱变化,尤其是在东亚人群中的特点,并未完全阐述清楚。通过研究中国II型糖尿病患者、糖尿病前期与糖耐受正常人群的血浆脂质代谢物组成及差异,可为亚洲人群的脂代谢研究提供参考。
实验设计:
样品:293个中国人的血浆样本,其中有114位II型糖尿病(T2D)患者,81位早期糖尿病患者(prediabetic),98位血糖正常的人(NGT)
研究方法:基于LC-MS/MS技术的脂质组学
实验结果:
脂质组检测共获得11,077 个特征峰,其中约46.77%可以匹配到一或多个脂质分子或脂类化合物。通过PERMANOVA 发现,脂质组数据和几个血浆参数以及生理情况密切相关。
共有1590 个峰在三个实验组中有显著差异,790 个可能匹配上脂质分子或脂类化合物。成对比较揭示出1269 个特征峰在NGT 和T2D 之间有显著差异,prediabetic 与NGT 和T2D 比较,分别有785 和578 个特征峰有显著差异,暗示着与NGT 相比,prediabetes 和T2D 拥有更为相似的脂质组。这些结果显示,从NGT 到prediabetes 再到T2D,代谢物会发生持续地变化。

图1-2 三组两两比较显著差异脂质的韦恩图;基于脂质全谱的随机树分类
主要结论:
1. 脂质组数据和几个血浆参数以及生理情况密切相关
2. 从NGT到prediabetes再到T2D,脂质会发生持续地变化
3. 成功构建28个特征峰模型,用于指示T2D的发生发展。AUC=90.23%
4. 揭示脂质分子和糖尿病的临床指标之间的相关性
案例二:脂质组学介导植物乙烯通路水解酶MHZ11功能的研究[2]
研究背景:
作为重要的植物激素,乙烯参与调节植物的生长、发育、应激反应等重要的生理过程。在拟南芥中,乙烯信号通路已经被揭示——乙烯和固定在内质网膜上的乙烯受体结合后,使受体转为非活性态,不能激活下游的CTR1,使得EIN2去磷酸化从而激活乙烯信号转导通路。但是在水稻中具体CTR1是如何调节乙烯信号通路的仍有待发现。
技术路线:
作者发现了一个水稻突变株mhz,其根部乙烯信号通路受到阻滞,随后通过分子生物学实验及脂质组学对关键蛋白MHZ11的底物及其参与到乙烯信号调节通路的调控规律进行研究。
主要结论:
1. 水稻突变株mhz11显示出根部对乙烯不敏感的特异性表型,基因分析发现其MHZ11基因发生,MHZ11作为正调控因子参与到水稻根部的乙烯信号调节通路中。
2. MHZ11是一个没有特异性的脂肪酶,通过脂质组学分析发现MHZ11可能参与磷脂的水解生成FFA,FFA可参与后续酯类的合成。

图3 水稻(野生型WT,突变株mhz11,MHZ11过表达株OE-4)根部的脂质表达统计图
3. MHZ11具有水解酶的活性,能够通过水解磷脂生成lyso-PE,FFA后与甾醇反应生成甾醇酯,膜表面甾醇含量的减少影响到乙烯受体及OsCTR1的相互作用,从而介导了下游乙烯信号通路的激活。

图4 MHZ11参与乙烯通路可能的过程图
参考文献
[1] Zhong, Huanzi, et al. "Lipidomic profiling reveals distinct differences in plasma lipid composition in healthy, prediabetic, and type 2 diabetic individuals." Gigascience 6.7 (2017): gix036.
[2] Zhao, He, et al. "The GDSL lipase MHZ11 modulates ethylene signaling in rice roots." The Plant Cell 32.5 (2020): 1626-1643.
1、数据预处理及质控分析
使用LipidSearch 软件进行LC-MS/MS数据处理,包括智能峰提取、脂质鉴定、峰对齐等的一系列分析。LipidSearch导出的数据通过metaX进行数据预处理、质控分析和后续分析。数据质控的内容包括,QC样本的BPC重叠图、所有样本的PCA分析、各组的CV分析。

图1 数据质控分析
2、鉴定及定量结果展示
(1)脂质分子数和亚类数的鉴定概况
国际脂质分类和命名委员会(International Lipid Classification and Nomenclature Committee)将脂类化合物分为八大类别(Category),每个类别又分为不同的大类(Main Class),每个大类可以根据极性头基的不同分为不同的亚类(Sub Class),每一亚类根据碳链不饱和度或长度等的差异分为不同的分子种属(Molecular Specie),每个分子种属对应多个不同的脂质分子(Lipid Molecule),构成脂类化合物的五级分类。对数据预处理后的脂质分子数量和亚类数量进行统计。
表1 脂质分子数量统计表
|
Lipid_Molecules_Number |
Sub_Classes_Number |
|
1208 |
36 |
(2)脂质亚类的鉴定及定量分析

图2 脂质亚类的鉴定和定量分析
3、差异脂质分子的筛选
通过PCA分析观察了解两组样本的差异情况。采用多变量分析PLS-DA模型前两个主成分的VIP值,结合单变量分析差异变化倍数(Fold Change, FC))和T检验(Student's t-test)的q-value值来筛选差异表达的脂质分子。筛选条件: 1)VIP ≥ 1;2) Fold-Change ≥1.2 或者 ≤ 0.8333;3) q-value < 0.05,同时满足这三个条件,即为差异脂质分子。

图3 差异脂质分子的筛选
4、差异脂质分析
包括差异脂质分子的聚类热图分析和差异脂质分子的相关性分析。

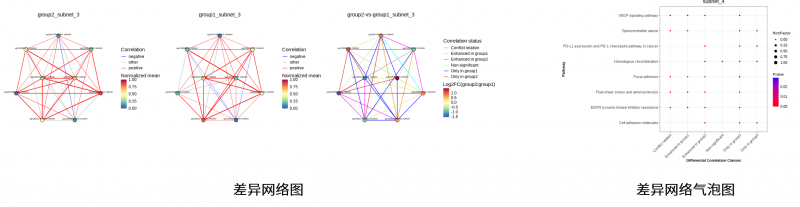
图4 差异脂质分子的分析
1、 重复次数要求
每组样本的生物学重复次数要求如下,重复次数越多越好。
表1 生物学重复次数要求
|
样本类型 |
重复次数要求 |
|
植物、微生物、细胞样本 |
≥ 6个 |
|
动物样本 |
≥ 10个 |
|
临床样本 |
≥ 30个 |
2、 送样量要求
表2 送样量要求
样本类型 | 建议送样量/例 | |
血清、血浆、尿液、血淋巴、脑脊液、房水 | ≥200 μL | |
滤纸条、拭子 | ≥4条 | |
外泌体(须是分离好的外泌体) | ≥500μL,约1011 Particles/mL | |
精浆、羊水、前列腺液、瘤胃液、呼吸冷凝液、胃灌洗液、肺泡灌洗液、汗液、唾液、痰液、牛奶、细胞培养液、发酵液 | ≥500 μL | |
动物及临床组织、粪便、肠道内容物、微生物菌体、脊髓、软骨 | ≥100 mg | |
细胞 | 107 个 (需每份样本细胞数目保持一样) | |
酒曲、酒醅、大曲发酵物等 | ≥500mg | |
土壤、淤泥、海水沉积物 | ≥500mg | |
植物样本 | 新鲜植物组织 | ≥500 mg |
冻干植物组织 | ≥ 200 mg | |
中药药汤 | 冻干粉≥200 mg,药液≥1mL | |
中药药丸 | ≥1g | |
Q1:为什么要使用质谱正负离子模式同时采集的方式进行分析?
A1: 不同的脂质在正(负)离子模式下电离的程度不同,如PC在正离子模式下更容易电离,而PE在负离子模式下更容易电离,同时采用正负离子模式对脂质组学进行数据采集,可以扩大脂质的覆盖度。
Q2:为什么要进行生物学重复?
A2:样本量少得到的结果个体差异可能会超越组间差异,使得结果可信度不高,甚至可能无法筛选出差异脂质 。因此对于不同的样本类型有不同的生物学重复次数要求。

