- 首页 > 全基因组Bisulfite甲基化测序
全基因组Bisulfite甲基化测序
产品简介
全基因组甲基化Bisulfite测序是DNA甲基化研究的黄金标准,它通过Bisulfite处理和全基因组DNA测序结合的方式,对整个基因组上的甲基化情况进行分析,具有单碱基的分辨率,可精确评估单个C碱基的甲基化水平,覆盖范围广。它可以构建精细甲基化图谱,建立表观遗传学研究数据库,为后续大规模开展不同样品间的甲基化差异分析提供参考图谱。
技术流程
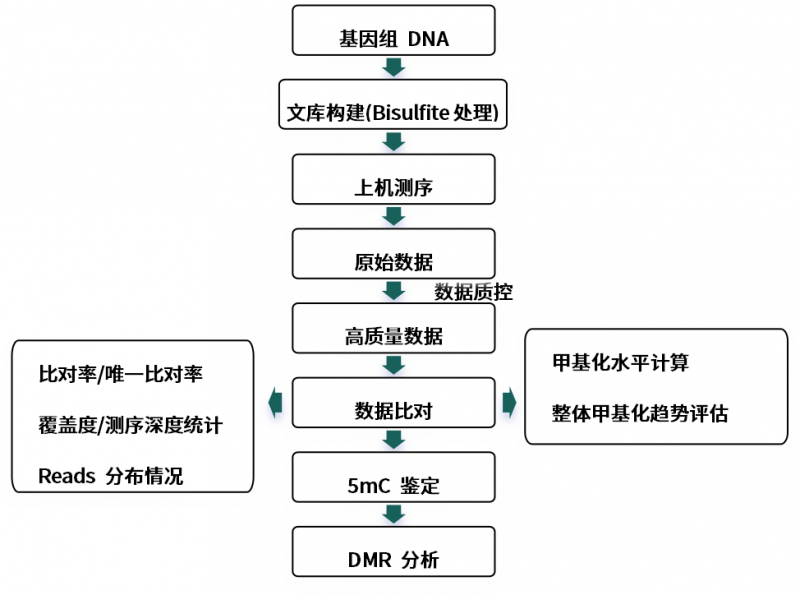
技术优势
技术稳定性、重复性好:从样本检测到文库构建、上机测序,每一步均有严格的标准操作SOP和对应的质控标准,保证每一个样本都得到真实的测序结果。
Bisulfite转化率高:经过不断优化建库方案,华大的甲基化建库BS平均转化率高达99%以上,作为文库构建的质控指标进行严格控制。
单碱基分辨率:精确分析每一个C碱基的甲基化状态。
准确性和可靠性高:准确区分甲基化的C和未甲基化的C。
检测范围广:覆盖全基因组范围内的所有位点。
无偏向性:反映真实的甲基化状态。
经验丰富:执行了上千个项目,数万个样本,合作发表文章一百多篇。
信息分析
表1 标准信息分析内容
信息分析条款 | 信息分析内容 |
标准信息分析 | 1. 数据基本处理与质控; 2. 比对结果统计; 3. 甲基位点统计与分析: 3.1. 甲基化位点鉴定; 3.2. 甲基化位点类型统计; 3.3. C位点甲基化水平分析(全基因组,染色体,功能元件); 3.4. 甲基化位点序列特征Motif图; 3.5. 甲基化水平IGV可视化文件(.bw); 3.6. 全基因组甲基化水平分布(样本/分组); 3.7. 基因基因上下游甲基化水平分布(样本/分组); 3.8. 样本相关性和PCA分析; 4. 差异甲基化分析: 4.1. 差异甲基化位点(DMC)鉴定; 4.2. DMC相关基因GO和 KEGG富集分析; 4.3. DMC锚定Promoter区域相关基因GO和KEGG富集分析; 4.4. 差异甲基化区域(DMR)鉴定; 4.5. DMR相关基因GO和KEGG富集分析; 4.6. DMR锚定Promoter区域相关基因GO和 KEGG富集分析。 |
Dr. Tom标准分析 | 1.数据基本处理与质控; 2.全基因组甲基化水平分析; 3.基因附近的甲基化水平分布; 4. TE附近的甲基化水平分布; 5.各染色体的平均甲基化水平; 6.基因组不同转录元件中的DNA平均甲基化水平; 7. mCG、mCHG和mCHH的甲基化胞嘧啶统计; 8.差异甲基化DMR的检测; 9.DMR相关基因的GO和Pathway分析; |
定制化信息分析 | 可结合客户的需求,协商确定定制化信息分析内容 |
文章题目:DNA damage triggers heritable alterations in DNA methylation patterns in Arabidopsis.
发表期刊:Molecular Plant(IF=24.1)
发表时间:2025年1月
研究技术:WGBS、RNA-Seq
研究结果:
DNA甲基化在维持基因组稳定性与调控基因表达方面发挥着关键作用,是动植物中动植物中高度保守的表观遗传机制。目前尚无足够的证据支持DNA损伤会导致全基因组范围内的DNA超甲基化。
本研究以积累单链 DNA 损伤的植物模型为研究对象,揭示 DNA 损伤显著提升基因组 DNA 甲基化水平,其影响与损伤强度及 DDR 通路密切相关。机制上,DNA损伤通过调控 DREAM 复合体增加对称性甲基化,通过 RdDM 途径增加非对称性甲基化。移除损伤或抑制 DDR 可逆转非对称性甲基化,但对称性 CG 甲基化稳定,具遗传记忆特性。研究表明 DNA 损伤是驱动植物基因组 DNA 甲基化水平及模式形成与演变的重要因素。
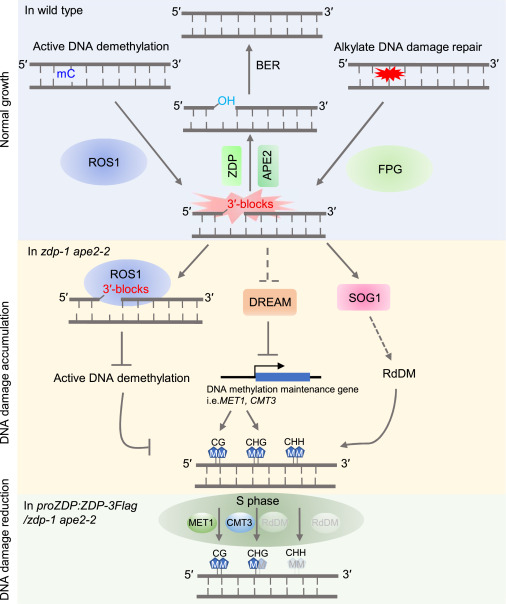
图1.DNA损伤驱动DNA甲基化的产生与演变的机制
案例二 DNA 甲基化应用于异染色质与维持基因组稳定性[2]
文章题目:H3T11 phosphorylation by CKII is required for heterochromatin formation in Neurospora.
发表期刊:Nucleic Acids Research(IF=13.1)
发表时间:2024年9月
研究技术:WGBS 、ChIP-Seq
研究结果:
异染色质是真核生物基因组的一个关键特征,对于维持基因组稳定性至关重要。然而,在丝状真菌粗糙脉孢菌(Neurospora crassa)中,导致重复诱导点突变遗迹处异染色质起始的机制尚不清楚,且独立于经典的RNAi途径。
本研究发现酪蛋白激酶II (CKII)及其激酶活性是在5H-cat-3区域明确的5kb异染色质上形成异染色质和其邻近的cat-3基因的转录抑制所必需的。催化亚基CKA与5H-cat-3区域内的H3T11磷酸化(H3pT11)共定位,CKA基因的缺失会导致H3T11磷酸化显著减少。此外,CKII激酶活性的丧失会导致H3pT11、H3K9me3和DNA甲基化水平显著降低,这表明CKII通过促进H3T11磷酸化来调节异染色质的形成。
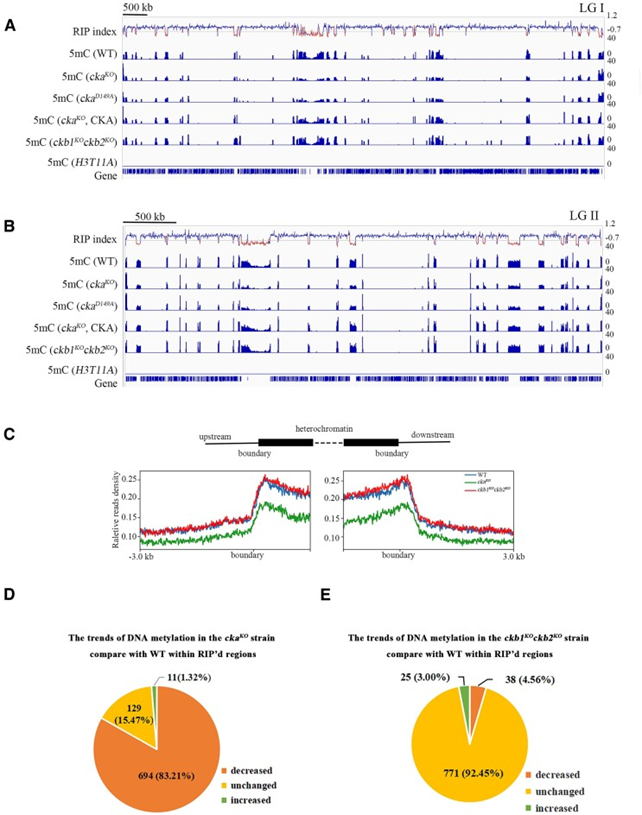
图 2 CKII在异染色质形成和DNA甲基化中的重要作用
案例三 TET1 双加氧酶为胰腺 β 细胞分化过程中 FOXA2 相关染色质重塑所必需(DNBSEQ WGBS) [3]
文章题目:TET1 dioxygenase is required for FOXA2-associated chromatin remodeling in pancreatic beta-cell differentiation.
发表期刊:Nature Communications(IF=15.7)
发表时间:2022年7月
研究技术:ATAC-seq、CMS-IP、WGBS、ChIP-Seq
研究结果:
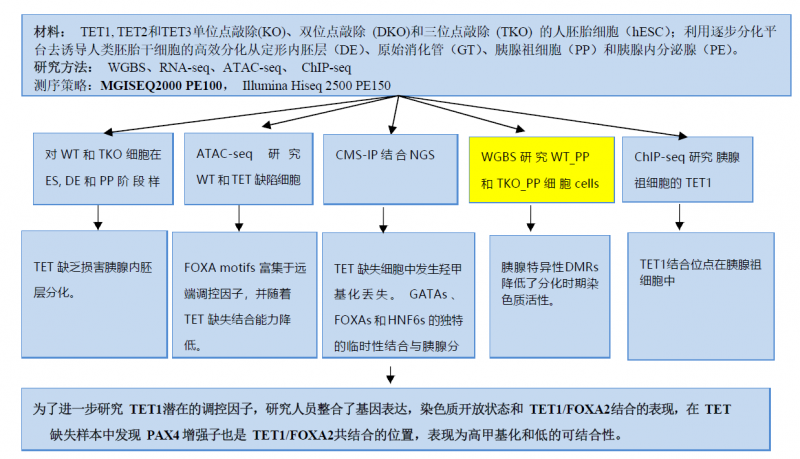
本研究综合利用全基因组分析鉴定独特的细胞类型特异性高甲基化区域(hyper-DMRS)显示出染色质活性降低并显著与FOXA2结合,而FOXA2是一个对胰腺分化很重要的先锋转录因子。
全基因组定位 TET1 显示 TET1 共定位于 FOXA2 靶点的一个子集,它在胰腺祖细胞中具有高水平的活性染色质修饰。结合这些发现和 TET1 失活后功能性 β 细胞的缺陷生成,该研究不仅揭示了 TET1 依赖的表观遗传调控在确定 β 细胞身份中的重要作用,而且为 TET 双加氧酶与先锋转录两者在谱系规范中的复杂互作关系提供了一个新的机制线索。
参考文献:
[1] Li J, Liang W, He XQ, Qian W. DNA damage triggers heritable alterations in DNA methylation patterns in Arabidopsis. Mol Plant. 2025 Mar 3;18(3):501-512.
[2] Tian Y, Zhang C, Tian X, et al. H3T11 phosphorylation by CKII is required for heterochromatin formation in Neurospora. Nucleic Acids Res. 2024 Sep 9;52(16):9536-9550.
Li J, Wu X, Ke J, et al. TET1 dioxygenase is required for FOXA2-associated chromatin remodeling in pancreatic beta-cell differentiation. Nat Commun. 2022 Jul 7;13(1):3907.
1. 甲基化位点水平分布
对于鉴定出的甲基化位点,计算其甲基化水平,公式为:ML=mC/(mC+umC)。其中ML为甲基化水平,并统计 不同类型的C碱基(mCG、mCHG和mCHH )的甲基化水平分布。
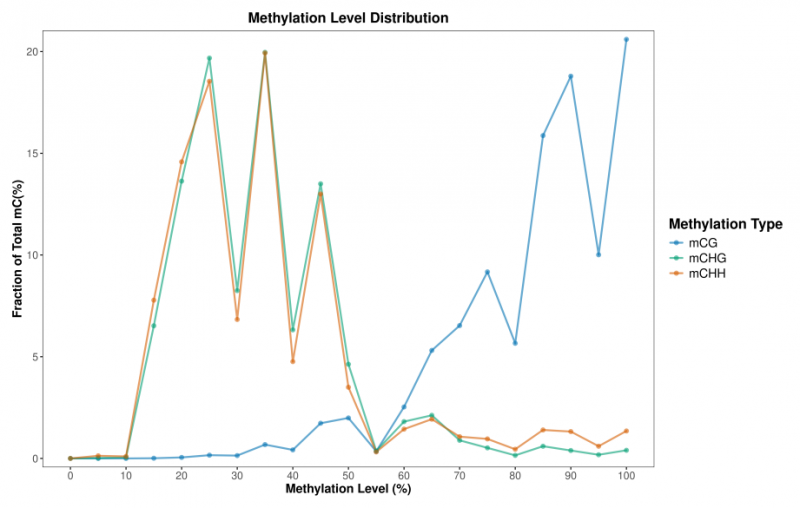
图1.样本甲基化位点水平分布图
2. 甲基化位点上下游特征序列 Motif 分析
Motif预测分析主要用于识别DNA、RNA或蛋白质序列中具有生物学功能的短保守序列模式,可以表征序列保守性以及可能在基因表达调控中发挥的作用。
本分析将在全基因组各位点类型( CG,CHG,CHH )中鉴定为甲基化的C位点及其上下游 9bp 的碱基序列作Logo Plots,研究不同序列环境中发生甲基化修饰的胞嘧啶上下游的序列特征。
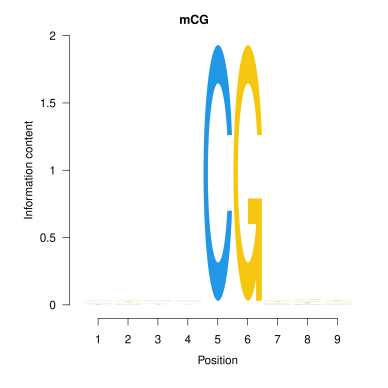
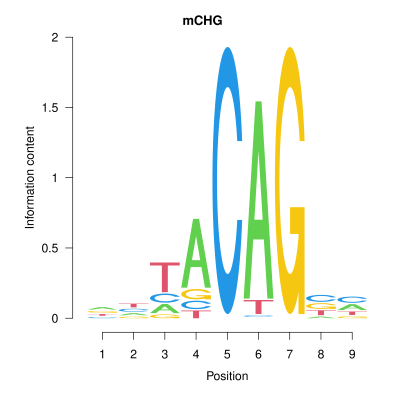
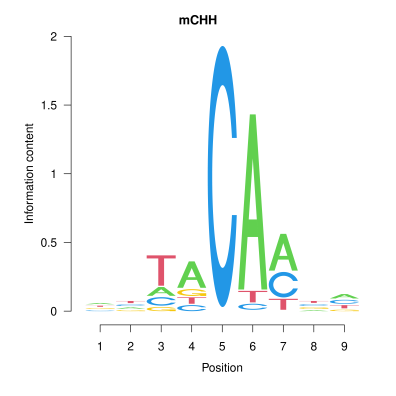
图2.甲基化C位点相邻碱基 Motif 图
3. 样本相关性与PCA分析
进行样本相关性与PCA分析。样品间甲基化水平的相关性是检验实验可靠性和样本选择是否合理的重要指标。相关系数越接近1,表明样品之间甲基化模式的相似度越高。
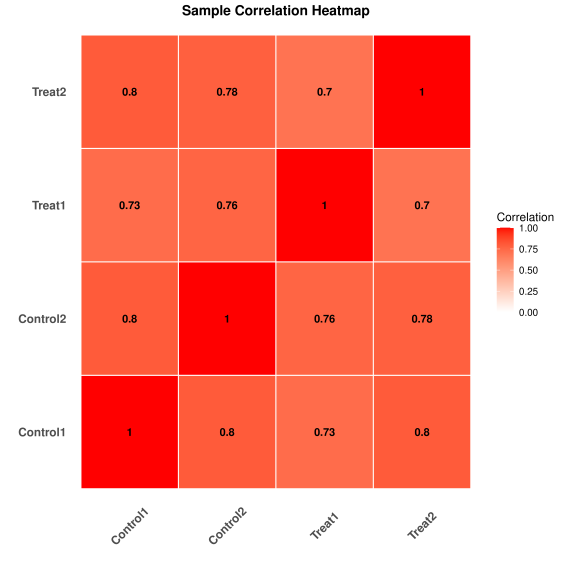
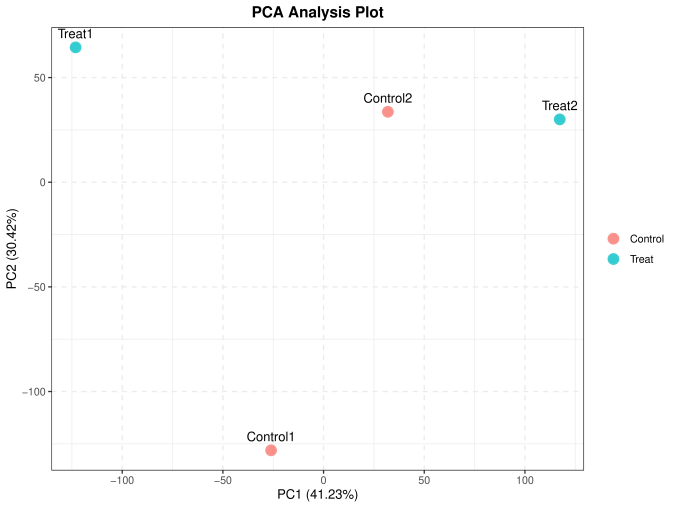
图3.样本相关性热图与PCA分析图
4. 差异性甲基化区域(DMR)相关基因GO分析
基因本体论(Gene ontology,GO)是所有物种中最主要的了解基因和基因产物属性的生物信息学分析手段,GO分析能够用于鉴定基因产物的性能,它包含了三类基因功能信息:细胞组分(Cellular Component),分子功能(Molecular Function)和生物学过程(Biological Process)。为了探讨表观遗传变异在通路和生物学过程中起到的作用,我们对DMR相关的基因进行了GO分析。
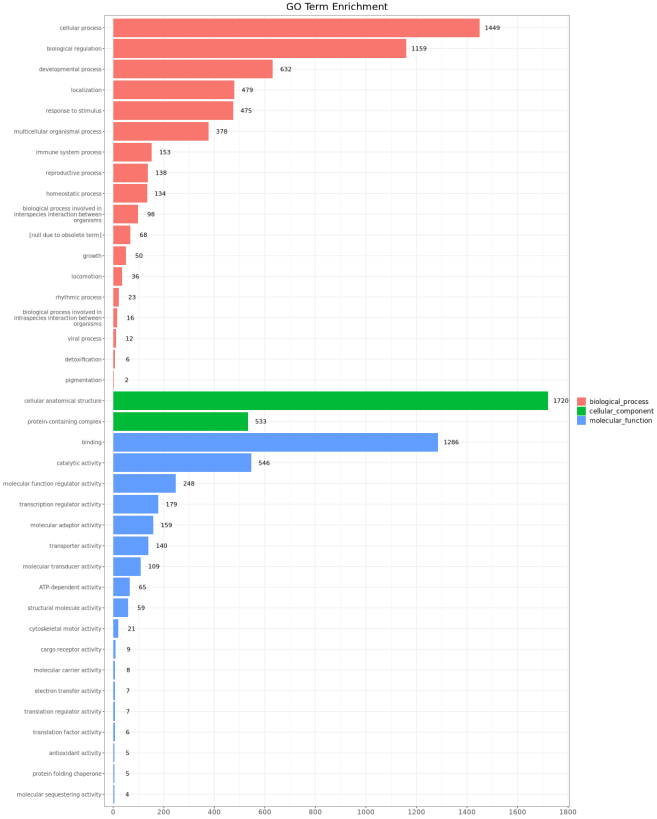
图4.DMR相关基因 GO 富集柱状图
5. 差异性甲基化区域(DMR)相关基因Pathway分析
KEGG (Kyoto Encyclopedia of Genes and Genomes)是有关Pathway的主要公共数据库,该数据库整合了基因组、化学以及系统功能信息,特别是测序得到的基因集与细胞、生物体以及生态环境的系统性功能相关联。所有样品中的DMR相关基因均用KEGG数据库进行分析。
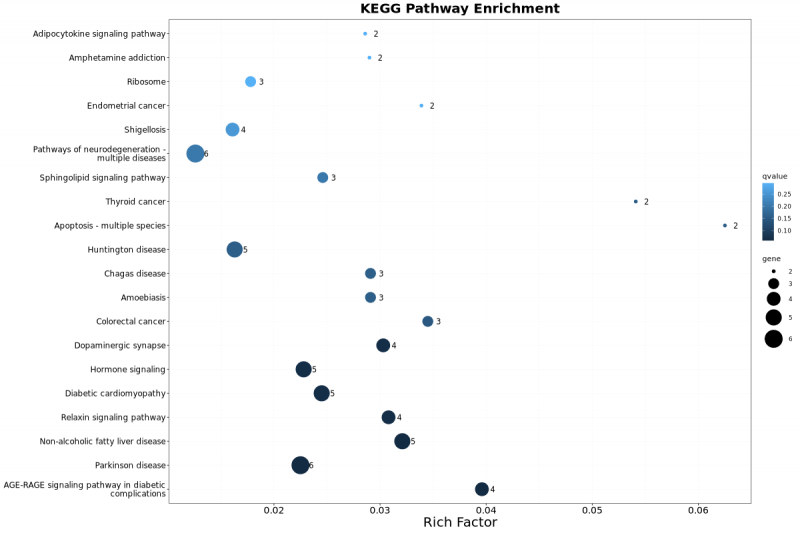
图5.DMR相关基因的Pathway功能显著性富集分析图
1、核酸样本
表1 DNA送样建议
样本类型 | 总量 | 浓度 | 完整性(胶图) | 纯度 |
Genomic DNA | ≥0.5μg | ≥12.5ng/μL | 主峰>20Kb | 无蛋白,RNA/盐离子等污染, 样本无色透明不粘稠 |
2、组织样本
表2 组织送样建议
组织类型 | 具体要求 |
新鲜培养细胞 (细胞数) | ≥1×106 cells |
新鲜动物组织干重 | ≥50 mg |
新鲜植物组织干重 | ≥200 mg |
全血(哺乳动物) | ≥0.6 mL |
全血(非哺乳动物) | ≥0.1 mL |
Q1: Bisulfite-Seq在项目开始之前需要考虑哪些因素?
A1:Bisulfite-Seq在项目开始之前需要考虑以下因素:是否为低甲基化率的物种;该物种的基因组完成情况如何(影响BS-SEQ的比对);基因组是否存在复杂因素:GC含量偏高、杂合度偏高、转座子、重复区域等。
Q2:可以对无参考基因组的物种进行Bisulfite研究吗?
A2:Bisulfite-Seq强烈依赖基因组的完成程度,基因质量的好坏直接影响后续的分析结果,因此更适合有完整基因组信息的物种。
Q3:如果合作伙伴自己建库,能否提供上机测序?如何进行文库质量检测并保证测序质量?
A3:合作伙伴自己建的文库,可以上机测序。如果用标准的Illumina kit,要注明kit的类型及货号。如果不是使用Illumina kit建库,那么合作伙伴提供信息单的同时必须说明建库方法、使用的试剂盒及品牌,以及提供建库所用的接头引物序列和预期文库片段大小。如果是index文库,要注明index的位置以及index的序列。若只完成部分建库过程请务必注明清楚;如果文库是PCR产物建库或者插入片段中有特定序列,请在提供的样品信息单中说明,否则会极大地影响数据质量。对合作伙伴文库的检测,首先用 Agilent 片段分析仪确定片段大小是否符合;然后通过Q-PCR精确定量确定上机体积。
Q4:Bisulfite的转化率是多少?
A4:Bisulfite转化率达到99%以上;如果样品的DNA不存在不发生甲基化的DNA作为对照,都会在样品中混入control DNA来验证Bisulfite的转化率。

